Imágenes de resonancia magnética en 3D como herramienta para el análisis de la calidad de lomo ibérico
La utilización de imágenes de resonancia magnética (MRI) junto con la aplicación de técnicas de Visión por computador y un análisis de datos adecuado, podría ser considerada como una metodología alternativa [13] al análisis físico-químico, debido a su naturaleza no destructiva, no invasiva, no intrusiva, no ionizante e inocua. Este procedimiento se lleva a cabo en tres etapas: la adquisición de imágenes MRI, el análisis de imágenes MRI y el tratamiento de los datos obtenidos.
Una vez adquiridas las imágenes MRI, éstas se analizan mediante algoritmos de visión por computador para la extracción de características de textura, con el fin de obtener datos numéricos que se procesarán posteriormente. Entre los algoritmos más empleados para el análisis de las imágenes se encuentran los algoritmos clásicos (2D), que son los que se han aplicado mayoritariamente para analizar imágenes MRI de productos cárnicos ([7], [8], [9], [11], [12]).
Los resultados obtenidos en estas investigaciones son razonablemente buenos, sin embargo, el estudio de las estructuras volumétricas (3D) podría suponer un paso adelante, ofreciendo nuevas posibilidades [10]. Esto es debido a que el mundo real no se basa en imágenes planas, sino tridimensionales. Teniendo esto en cuenta, el uso de imágenes 2D supondría una pérdida de información, mientras que trabajar con imágenes 3D significaría tratar de obtener toda la información contenida dentro de las imágenes. Los estudios centrados en imágenes 3D están suscitando gran interés, sobre todo en el campo de la medicina, principalmente para la detección y clasificación de tumores [2]. En productos cárnicos, se han encontrado pocos ejemplos de trabajos con imágenes en 3D [4]. En 2015 se propuso un nuevo algoritmo 3D para estudiar la distribución de texturas en imágenes de lomos tridimensionales desde diferentes orientaciones [5]. La aplicación de este algoritmo ha permitido determinar algunos atributos sensoriales del lomo de forma no destructiva [5].
Una vez analizadas las imágenes, el último paso consiste en estudiar los datos numéricos proporcionados por los algoritmos de visión por computador. Normalmente se emplean herramientas estadísticas, como el coeficiente de correlación de Pearson y el análisis de componentes principales, empleando técnicas de validación optimistas.
El objetivo de este trabajo es predecir los parámetros físico-químicos de lomos ibéricos a través de reconstrucciones 3D MRI.
Diseño experimental
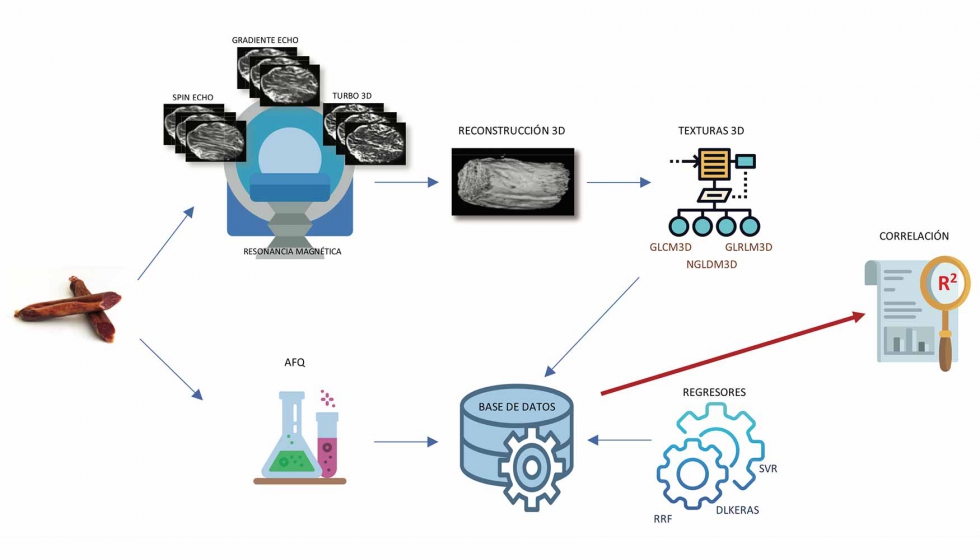
La figura 1 muestra el diseño experimental llevado a cabo en este trabajo. En primer lugar, se obtuvieron las imágenes MRI de lomos Ibéricos (frescos (n=5) y curados (n=5)) aplicando 3 secuencias de adquisición (Spin Echo (SE), Gradiente Echo (GE) y Turbo 3D (T3D)), utilizando un dispositivo de MRI de bajo campo. Una vez obtenidas las imágenes, se realizaron análisis físico-químicos (destructivos) de los lomos para determinar el porcentaje de humedad, el contenido de agua, el color instrumental (L, a* y b*) y el porcentaje de grasa en extracto húmedo y seco.
El análisis de las imágenes comenzó con un proceso de interpolación lineal seguido de un proceso de reconstrucción 3D.
Todas las reconstrucciones 3D MRI fueron analizadas mediante 3 métodos de textura 3D, obteniéndose un vector de características de textura computacional para cada una de ellas. A continuación, se construyó una base de datos con los resultados del análisis de las reconstrucciones y de los análisis físico-químicos. Sobre esta base de datos se aplicaron 28 regresores avanzados para obtener ecuaciones de predicción de los parámetros de calidad del lomo en función de las características computacionales. Así, en este estudio se obtuvieron 252 ecuaciones para cada parámetro físico-químico analizado, como consecuencia del empleo de 3 secuencias de adquisición, 3 métodos de textura 3D y 28 regresores (3x3x28). Los resultados de predicción obtenidos se evaluaron mediante una variante de validación cruzada y usando el coeficiente de determinación R2. Por último, se aplicó el Ranking de Freedman.
Materiales
Como materiales se utilizaron lomos frescos y curados escaneados mediante el dispositivo de resonancia de bajo campo VET-MR E-SCAN XQ (figura 2), con un imán de 0.18 T, el cual se encuentra localizado en el Servicio de Análisis e Innovación en Productos de Origen Animal (SiPA) de la Universidad de Extremadura.

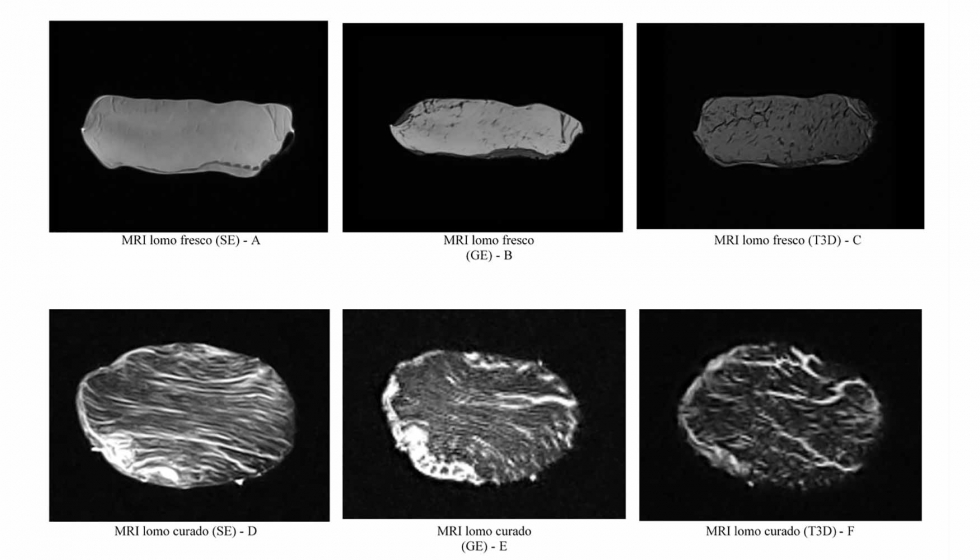
Para la obtención de las imágenes se emplearon tres tipos de secuencias de adquisición: Spin Echo (SE), Gradient Echo (GE) y Turbo 3D (T3D), las cuales se potenciaron en T1 como tiempo de relajación (figura 3).
Metodología
Análisis físico-químico
Las características físico-químicas que se determinaron en este trabajo son, el porcentaje de humedad, el contenido de agua, el color instrumental (L, a* y b*) y el porcentaje de grasa en extracto húmedo y seco.
Reconstrucción 3D
Con las imágenes obtenidas con cada secuencia de adquisición (29, 29 y 122, para SE, GE y T3D, respectivamente) se llevó a cabo un proceso de interpolación lineal para cubrir el hueco existente entre cada dos imágenes consecutivas, completándose con el proceso de reconstrucción 3D mediante VTK (Visualization Toolkit), librería multiplataforma para manipular y visualizar datos científicos. La figura 4 muestra un ejemplo de lomo ibérico curado reconstruido en 3D.

Análisis computacional de las MRI
Sobre las reconstrucciones 3D de lomo se aplicaron tres algoritmos de textura tridimensionales [6] (adaptaciones desarrolladas de algoritmos clásicos de textura 2D): GLCM3D (matriz de coocurrencia de niveles de gris 3D), NGLDM3D (matriz de dependencia de niveles de gris en un vecindario 3D) y GLRLM3D (matriz de longitud de hileras de niveles de gris 3D.
Análisis de datos
Sobre la base de datos compuesta por los datos computaciones y físico-químicos, se aplicaron 28 regresores [14]. Así, se obtuvieron ecuaciones de predicción para cada parámetro de calidad en función de estas características computacionales. La predicción se certificó mediante un método de validación cruzada realista basado en 3 conjuntos: entrenamiento, validación y test. Finalmente, para evaluar la bondad de las ecuaciones obtenidas, se calculó el coeficiente de determinación R2. Los resultados de predicción también fueron analizados mediante el Ranking de Freedman, comparando las diferentes combinaciones de los métodos empleados.
Resultados
Para cada combinación compuesta por la secuencia de adquisición MRI (3), método de textura 3D (3) y regresor (28), se obtuvo la correspondiente ecuación de predicción (252) para cada uno de los parámetros físico-químicos evaluados. La tabla 1 muestra la mejor combinación de secuencia de adquisición, método de textura y regresor para predecir cada uno de los parámetros físico-químicos analizados. Para todos los casos, la R2 es superior a 0.56, lo que indica que las predicciones son de muy buenas a excelentes.
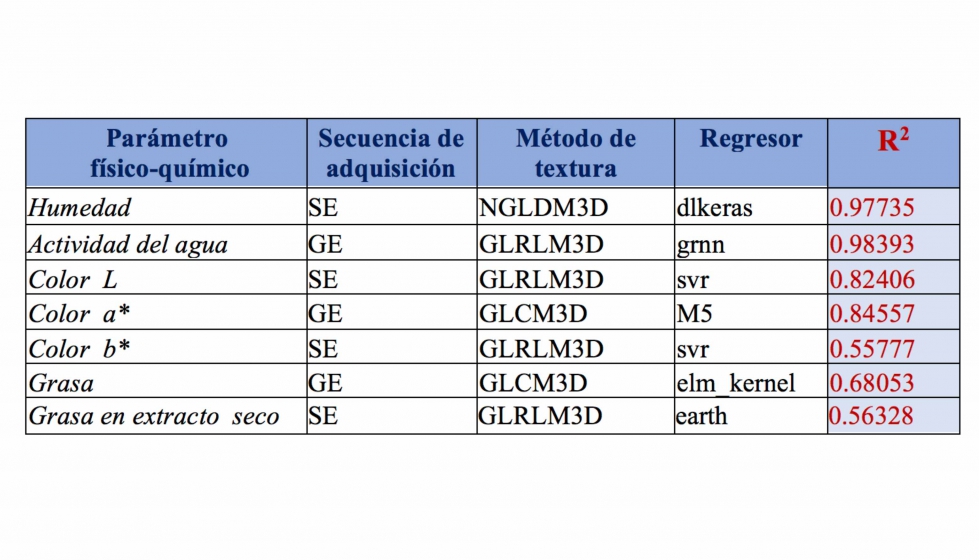
No obstante, como se puede observar en la tabla anterior, no existe una combinación común que permita predecir todas las características (parámetros físico-químicos). Para ello, se aplicó el Ranking de Freedman, obteniéndose como resultado que la mejor combinación es SE-GLRLM3D- elm_kernel. Esta combinación permite predecir 6 de las 7 características con un coeficiente de correlación aceptable.
Conclusiones
El análisis de reconstrucciones 3D MRI de lomo obtenidas tras un proceso de interpolación y reconstrucción en 3D puede llevarse a cabo mediante la adaptación a 3D de los algoritmos clásicos de textura.
Se pueden predecir las características físico-químicas del lomo ibérico a partir de reconstrucciones 3D MRI aplicando diferentes combinaciones de secuencia de adquisición-método de textura 3D-regresor, pudiéndose indicar el empleo de SE-GLRLM3D-elm kernel para predecir 6 de las 7 características físico-químicas de forma fiable.
Referencias bibliográficas
- [1] Alasvand, S., Kadivar, M., Aminlari, M., Shekarforoush, S., 2012. A comparative study of physico-chemical and functional properties, and ultrastructure of ostrich meat and beef during aging. CyTA - J. Food 10 (3), 201-209.
- [2] Arunadevi, B., Nachimuthu, S., 2013. Texture analysis for 3D classification of brain tumor tissues. Przeglad Elektrotechniczny 89, 338-342.
- [3] Ávila, M. M. 2018. Evaluación de técnicas avanzadas de regresión y de características de textura en imágenes de resonancia magnética para determinar parámetros de calidad en productos cárnicos. Tesis doctoral. Universidad de Extremadura.
- [4] Ávila, M.M., Durán, M.L., Antequera, T., Palacios, R., Luquero, M., 2007. 3D reconstruction on MRI to analyse marbling and fat level in Iberian loin. Lect. Notes Comput. Sci. 4477, 145-152.
- [5] Ávila, M.M., Caballero, D., Durán, M.L., Caro, A., Pérez-Palacios, T., Antequera, T., 2015b. Including 3D-textures in a computer vision system to analyze quality traits of loin. Lect. Notes Comput. Sci. 9163, 456-465.
- [6] Ávila, M. M., Caballero, D., Antequera, T., Durán, M. L., Caro, A. and Pérez-Palacios, T. Applying 3D texture algorithms on MRI to evaluate quality traits of loin. Journal of Food Engineering, 222:258–266, 2018.
- [7] Caballero, D., Antequera, T., Caro, A., Durán, M.L., Pérez-Palacios, T., 2016a. Data Mining on MRI-Computational texture features to predict sensory characteristics in ham. Food Bioprocess Technol. 9, 699-708.
- [8] Caballero, D., Caro, A., Rodríguez, P.G., Durán, M.L., Ávila, M.M., Palacios, R., Antequera, T., Pérez-Palacios, T., 2016b. Modeling salt diffusion in Iberian ham by applying MRI and data mining. J. Food Eng. 189, 115-122.
- [9] Cernadas, E., Carrión, P., Rodríguez, P.G., Muriel, E., Antequera, T., 2005. Analyzing magnetic resonance images of Iberian pork loin to predict its sensorial characteristics. Comput. Vis. Image Underst. 98, 345-361.
- [10] Melado-Herreros, A., Muñoz-García, M.A., Blanco, A., Val, J., Fernández-Valle, M.E., Barreiro, P., 2013. Assessment of watercore development in apples with MRI: effect of fruit location in the canopy. Postharvest Biol. Technol. 86, 125-133.
- [11] Pérez-Palacios, T., Antequera, T., Durán, M.L., Caro, A., Rodríguez, P.G., Palacios, R., 2011a. MRI-based analysis of feeding background effect on fresh Iberian ham. Food Chem. 126, 1366-1372.
- [12] Pérez-Palacios, T., Ruiz, J., Martín, D., Barat, J.M., Antequera, T., 2011b. Pre-cure freezing effect on physiochemical, texture and sensory characteristics of Iberian ham. Food Sci. Technol. Int. 17, 127-133.
- [13] Pérez-Palacios, T., Caballero, D., Antequera, T., Durán, M. L., Ávila, M. M., and Caro, A. 2017. Optimization of MRI Acquisition and Texture Analysis to Predict Physico-chemical Parameters of Loins by Data Mining. Food Bioprocess Technology, 10:750–758.
- [14] Sirsat, M. S. Application of machine learning to agricultural soil data. 2017. PhD thesis. Universidad de Santiago de Compostela (CiTIUS).