Expresión diferencial de genes en cerdo Ibérico de montanera divergentes para mioglobina
Miguel Ángel Fernández-Barroso1, Juan María García-Casco1,2, Yolanda Núñez Moreno2, Luisa Ramírez-Hidalgo3, Alberto Márquez3, Patricia Palma-Granados1,2 y María Muñoz2
1Centro de I+D en Cerdo Ibérico, INIA-CSIC, Ctra Ex101 km4,7 06300 Zafra (Badajoz)
2Dpto. Mejora Genética Animal, INIA-CSIC, Ctra La Coruña km 7,5, 28040 Madrid
3Sánchez Romero Carvajal S.A., Ctra San Juan del Puerto s/n, 21290 Jabugo (Huelva)
07/11/2023El cerdo ibérico se distingue por producir una carne y unos productos curados de calidad excepcional. El color rojo brillante de la carne fresca es crucial en la elección de los consumidores, ya que es reflejo de su calidad, y este color está directamente relacionado con la presencia de mioglobina, una proteína parecida a la hemoglobina que se encuentra en el músculo. El propósito de este estudio fue identificar posibles genes candidatos que puedan influir en el contenido en mioglobina, con el objetivo de incorporarlos en programas de mejora genética para el cerdo Ibérico.

El cerdo Ibérico se caracteriza por su carne de gran calidad y sus apreciados productos curados. Esta calidad está asociada a las particularidades genéticas de la raza (Alves et al., 2003), con un enorme apetito, un alto potencial graso e índice de recambio proteico, y una baja deposición de tejido magro (Rivera-Ferre et al., 2005), que a su vez están determinadas por su singular sistema tradicional de producción al aire libre. El color es una de las principales características de la calidad de la carne, y es la primera que el consumidor percibe sensorialmente. Por ello, es un factor importante de decisión en el momento de la compra de carne para su consumo en fresco. El color rojo cereza brillante normalmente se usa como un indicador saludable en la carne fresca, mientras que las que son demasiado oscuras o pálidas no suelen satisfacer las preferencias del consumidor.
La mioglobina (MB), proteína responsable del transporte de oxígeno en músculo, es el principal pigmento relacionado con el color rojo de la carne (Suman & Poulson, 2013). Cuando la mioglobina se une al oxígeno de manera reversible (oximioglobina) le da a la carne un color rojo cereza, fundamental para la aceptación del consumidor. La mioglobina se puede encontrar en otros estados, proporcionando distintos colores: la deoximioglobina que confiere un color rojo más oscuro o la metmioglobina, que le da un color marrón. Todas estas formas pueden ser cuantificadas mediante espectrofotometría.
El contenido de mioglobina en la carne está influenciado por diferentes factores como la especie, la raza, el tipo de músculo, la edad y el sistema de producción. De hecho, varios autores han señalado que la carne de cerdos criados en sistemas extensivos al aire libre tiene un mayor contenido en mioglobina y son más rojas (Ventanas et al., 2005, Pugliese et al., 2005). Desde un punto de vista genético, es un carácter moderadamente heredable, por lo que puede ser incluido en un programa de selección. Además, el contenido en MB está altamente correlacionado genéticamente con el color rojo a* medido por colorímetro, por lo que seleccionando para uno de los caracteres se puede seleccionar el otro (Fernández-Barroso et al., 2020a).
La inclusión del contenido de MB como objetivo de selección en programas de mejora genética requiere de su determinación analítica, que puede resultar compleja y laboriosa, por lo que una alternativa podría ser la utilización de selección asistida por marcadores genéticos, mediante la cual la selección de una variante génica asociada favorablemente al contenido en MB permitiría mejorar este carácter. En un trabajo previo, hemos detectado algunos marcadores genéticos asociados al carácter que están localizados en genes candidatos como CASP9 y PRKAG3 (Fernández-Barroso et al., 2020a).
Uno de los métodos para identificar genes candidatos para un carácter en particular consiste en la secuenciación completa del transcriptoma de grupos de individuos divergentes para el mismo. El transcriptoma es el conjunto de genes que se expresa en un tejido o célula en un momento concreto del tiempo, esta expresión es dinámica y cambia en función del tejido, la edad y de diferentes factores ambientales como la dieta. La detección de genes con expresión opuesta en estos grupos divergentes puede explicar la expresión fenotípica del carácter, por lo que esta metodología permite una mejor comprensión de los mecanismos biológicos subyacentes. Esta estrategia la hemos aplicado en trabajos anteriores, mediante la técnica de secuenciación del transcriptoma denominada RNA-seq, en la búsqueda de genes candidatos para el porcentaje de grasa intramuscular (Muñoz et al., 2018) y para la terneza de la carne (Fernandez-Barroso et al., 2020b).
Por ello, en este trabajo, secuenciamos el transcriptoma del lomo en cerdos ibéricos divergentes para el contenido en MB. Los objetivos globales planteados en el estudio fueron identificar y cuantificar todos los genes expresados diferencialmente y llevar a cabo análisis funcionales de las vías biológicas que podrían estar involucradas en las diferencias en el contenido en MB.
Materiales y métodos
Animales y datos fenotípicos
El manejo de los animales se realizó de acuerdo con las normas de la Política Española de Protección Animal RD 53/2013, que da cumplimiento a la Directiva de la Unión Europea 2010/63/UE sobre la protección de los animales utilizados en investigación. Los cerdos Ibéricos utilizados en este estudio fueron 828 machos castrados de tres campañas de montanera pertenecientes a la línea SRC de Sánchez Romero Carvajal. Su manejo y alimentación fue el habitual de este tipo de sistema de producción. Después del sacrificio, que se realizó con una edad aproximada de 17 meses y 165 kg de peso final medio, se tomaron porciones de lomo de cada individuo y se almacenaron a -20 °C hasta la determinación del contenido en MB. En el momento del análisis las muestras se descongelaron y se midió MB como mg de mioglobina/g de músculo. El contenido medio de MB fue de 1,77 mg/g, con una desviación típica de 0,31.
Una vez conocido el contenido de MB de los 828 animales, se realizó una evaluación genética para MB siguiendo la metodología habitual BLUP-Modelo Animal que se utiliza en los programas de mejora convencionales. En el modelo estadístico se incluyó el porcentaje de grasa intramuscular, la edad de sacrificio y el peso promedio de los dos lomos de cada animal como covariables. Además, como efectos aleatorios se incluyeron el efecto genético aditivo con la matriz de pedigrí de todos los animales disponibles en la genealogía, y los efectos ambientales debidos al lote de montanera y a la fecha de sacrificio. Con este modelo se estimaron los valores genéticos de mejora (VGM) utilizando los programas PEST 4.1 y vce-6 (Groeneveld et al., 1999 y 2010). Se seleccionaron 12 cerdos con los VGM más extremos para MB, seis por cada grupo, evitando hermanos completos. Los animales con VGM más extremos pertenecían a la misma campaña, por lo que se redujeron los posibles efectos ambientales anuales que se asocian a las montaneras. La tabla 1 muestra las medias del contenido en MB y de los valores estimados de VGM en los dos grupos, MB Alto y MB Bajo.
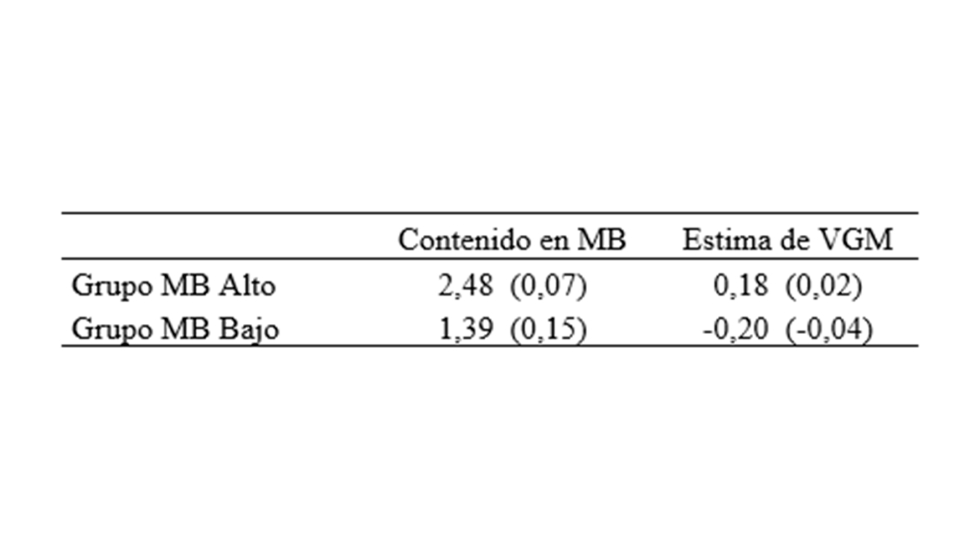
Tabla 1. Media y desviación típica (entre paréntesis) del contenido y de las estimas del valor genético de mejora en mioglobina (MB) en los grupos de cerdos divergentes.
Análisis del transcriptoma
Para el análisis del transcriptoma se tomaron muestras de lomo y se almacenaron en tubos criogénicos, los cuales fueron congelados de inmediato en nitrógeno líquido tras el sacrificio. Estas muestras se conservaron a -80 °C hasta su posterior análisis. Para la extracción de ARN total se utilizó un kit comercial de purificación de ARN de alta calidad (RiboPure™, Ambion). El ARN se cuantificó y se valoró su integridad obteniéndose en todas las muestras valores superiores al mínimo exigido para aplicar esta técnica. Se construyeron las librerías para cada muestra y éstas se secuenciaron en un analizador de secuencias Novaseq 6000 de Illumina.
Los análisis bioinformáticos se llevaron a cabo utilizado diferentes programas y herramientas informáticas según se muestra en la figura 1. Para decidir si un gen estaba diferencialmente expresado (GDE) se aplicaron criterios basados en el denominado fold change. Cuando las diferencias de expresión entre los grupos de MB Alto y MB Bajo fueron inferiores a -0,58 o superiores a 0,58, con un valor de q menor a 0,10, se consideraron GDE. Los análisis funcionales de los GDE encontrados se realizaron mediante la información de la denominada ontología génica (GO), utilizando el navegador FatiGO y el progama String Tools que permiten una interpretación biológica de los datos.

Figura 1. Esquema de los programas utilizados en el análisis bioinformático.
Los resultados del análisis RNA-seq se validaron mediante la técnica de RT-qPCR. Todos las referencias a estos métodos y herramientas pueden ser consultadas en Fernández-Barroso et al. (2022).
Resultados y discusión
Todas las muestras analizadas pasaron los controles de calidad establecidos y entre el 91,8 % y el 94,1 % de las secuencias leídas alinearon correctamente con el genoma de referencia porcino (Sscrofa11.1; tabla 2). Es decir, la reconstrucción del transcriptoma tuvo éxito en las muestras puesto que un porcentaje muy elevado, superior al 90%, puede ser reconocido en el genoma porcino.
De un total de 22.452 genes anotados en el genoma de referencia, se han detectado 16.746 expresándose en nuestras muestras. El gráfico del tipo volcano plot (figura 2) muestra todos los genes expresándose en el conjunto de muestras, en el cual los puntos rojos muestran los genes GDE. Se identificaron un total 57 GDE entre los grupos MB Alto y MB Bajo, de los cuales 53 GDE tenían mayor expresión en el grupo MB Alto (log2 FC ≥ 0.58), mientras que los otros cuatro mostraron mayor expresión en el grupo MB Bajo (log2 FC ≤ −0.58). El análisis de agrupamiento jerárquico utilizando los GDE, dividió correctamente las muestras en los grupos MB Alto y MB Bajo, como se aprecia en la figura 3.
Los valores de expresión, log2 FC, variaron entre −1.26 a 5.42, siendo los valores extremos para los genes que codifican para las enzimas carbohidrato sulfotransferasa 1 (CHST1, log2 FC = 5.42, p = 2.16×10−5, mayor expresión en el grupo MB Alto) y enoil-CoA hidratasa 1 (ECH1, log2 FC = −1.26, p = 1.33 × 10−4, mayor expresión en el grupo MB Bajo). La tabla 2 muestra una lista de los GDE elegidos por sus funciones clave, que estarían asociadas con las vías biológicas del contenido en MB.
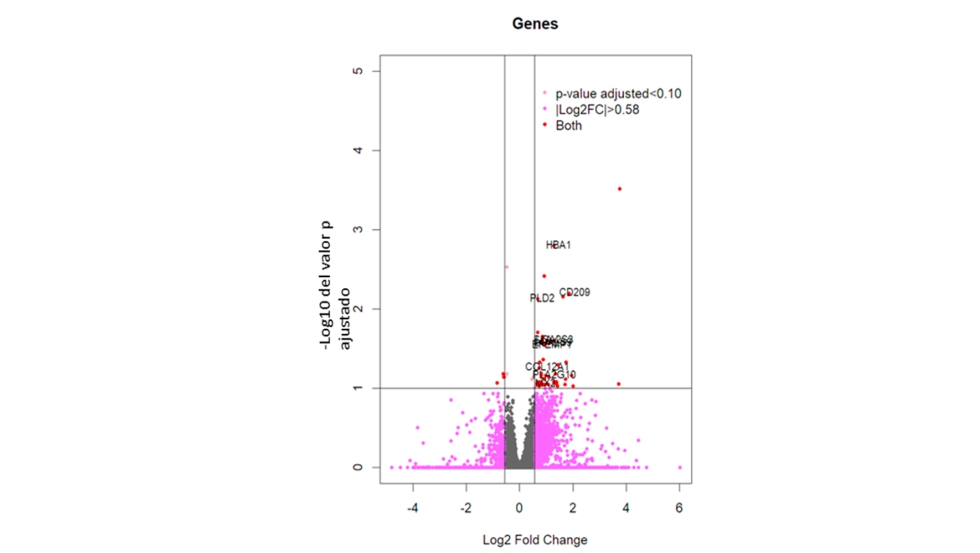
Figura 2. Gráfico volcano plot con los genes diferencialmente expresados (puntos rojos).
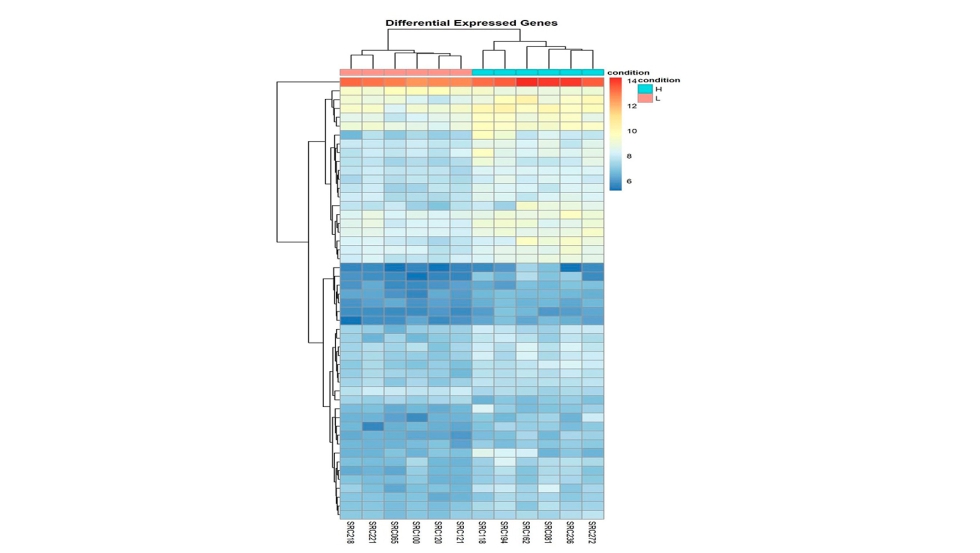
Figura 3. Análisis de agrupamiento jerarquizado de las muestras de Alto o Bajo contenido en mioglobina en función de los genes diferencialmente expresados.
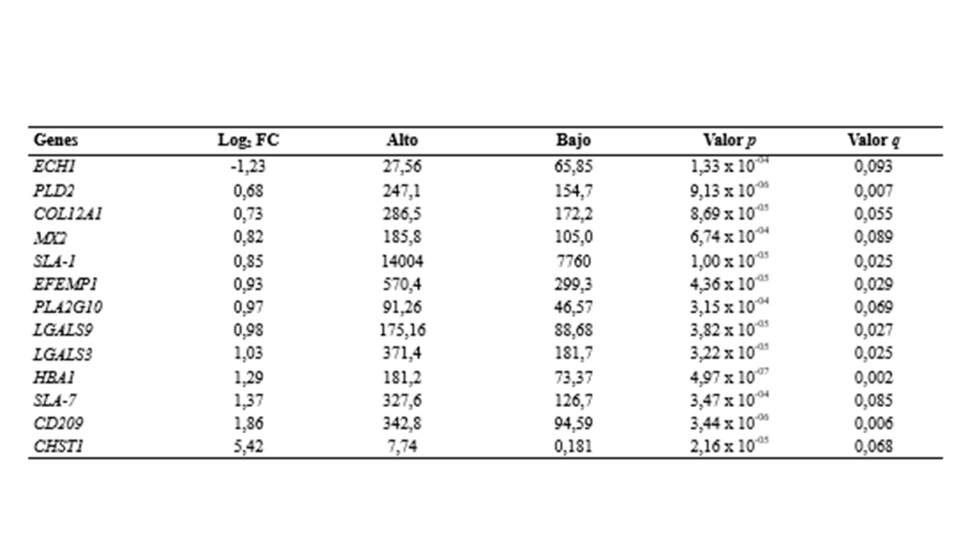
Tabla 2. Fold change (FC), valor medio de expresión en los grupos Alto y Bajo en mioglobina y valores p y q correspondientes a los genes más relevantes diferencialmente expresados (GDE).
Análisis funcionales
Los análisis de geontología realizados con la herramienta FatiGO reconocieron diversos procesos biológicos en los que intervenían los GDE (tabla 3). Estos procesos estaban implicados en el metabolismo de las prostaglandinas, el metabolismo de los eicosanoides, el estado de oxidación, el transporte de ácidos grasos, el metabolismo de las especies reactivas de oxígeno, la proliferación de células T, la organización del citoesqueleto y el tejido conectivo.

Tabla 3. Lista de términos de ontología génica relacionados con el contenido en mioglobina en los genes diferencialmente expresados usando FatiGO.
Finalmente, la figura 4 muestra una red de interacciones proteína-proteína codificadas por los GDE identificados. Encontramos cuatro grupos diferenciados, reguladas positivamente en el grupo MB Alto. El grupo 1 está involucrado en el reconocimiento celular y molecular y en el metabolismo inmunológico, el grupo 2 en la defensa contra virus, el grupo 3 está asociado con la unión de actina y la organización de la estructura de actomiosina y citoesqueleto y el grupo 4 con la función celular, la adhesión celular y la degradación de la matriz extracelular.
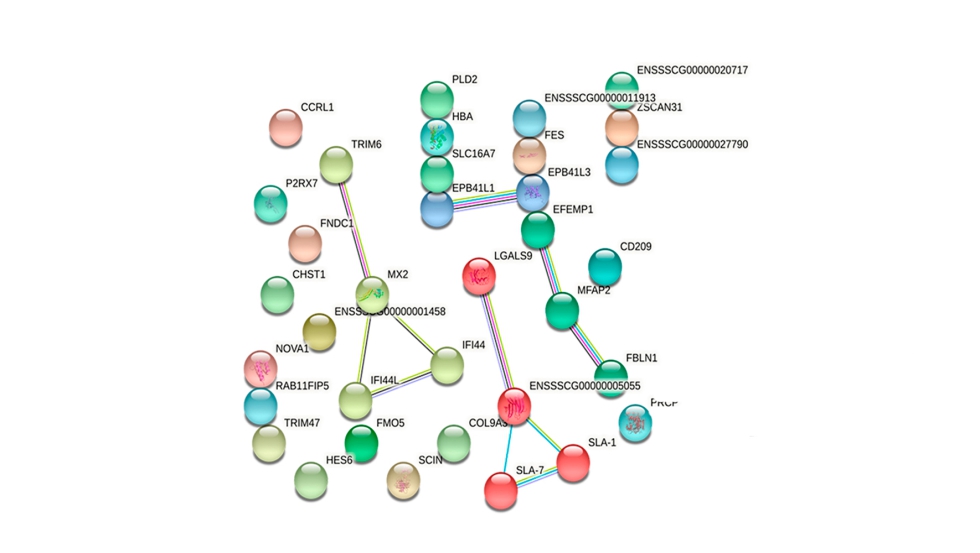
Figura 4. Red de interacciones proteína-proteína construida a partir de los GDE. Los nódulos del mismo color que comparten líneas múltiples están agrupados en el mismo clúster.
Del conjunto de genes diferencialmente expresados, nos centramos en este artículo en los que están claramente relacionados con el metabolismo lipídico del animal. En este caso, podemos destacar PLA2G10 (fosfolipasa A2 del grupo X) y PLD2 (fosfolipasa D2), ambas con mayor expresión en el grupo de MB Alto, y ECH1 (dienoil-CoA isomerasa mitocondrial), con mayor expresión en el grupo de MB Bajo.
El gen PLA2G10 codifica una enzima participa en la hidrólisis de los fosfolípidos de la membrana celular y en la liberación de ácidos grasos y lisofosfolípidos (Murakami et al., 2020). Según el análisis funcional, este gen está implicado en la regulación de la secreción y transporte de prostaglandinas, transporte de ácidos grasos y regulación de la secreción de eicosanoides. La enzima tiene un papel en el mantenimiento de la homeostasis de los fosfolípidos de la membrana. Además, desempeña una función significativa en procesos inflamatorios al liberar ácido araquidónico, un precursor de los eicosanoides. La proteína PLA2G10 podría estar involucrada en funciones inmunes y también actúa como un importante mecanismo de defensa contra parásitos y virus intestinales, con un papel en las respuestas inmunes adaptativas e innatas (Murakami et al., 2020). Por tanto, este gen realizaría a la vez una doble función clave: el metabolismo lipídico y el inmunológico.
El gen PLD2 codifica una enzima que desempeña un papel fundamental en la regulación de la función y el destino celular. El análisis funcional reveló que este gen está involucrado en la vía de biosíntesis III de colina. Además, PLD2 hidroliza la fosfatidilcolina de la membrana celular generando ácido fosfatídico, que es un mensajero lipídico que media en las funciones de señalización y que está involucrado en la respuesta sináptica inducida por hierro (Mateos et al., 2012). Cabe destacar que la mioglobina es uno de los principales depósitos de hierro en los mamíferos. Por tanto, un mayor contenido de mioglobina podría estar relacionado con una mayor liberación de hierro y, en consecuencia, una mayor activación de PLD2.
El gen ECH1 es uno de los pocos menos expresados en el grupo MB Alto. Este gen codifica una enzima que hidrata la enoil-CoA de cadena corta y media y está relacionado con la regulación positiva de la β-oxidación de ácidos grasos (Bahnson et al., 2002).
En general, los resultados que se obtuvieron en este trabajo indican que los cerdos ibéricos con un contenido alto en MB muestran una mayor expresión de los genes que codifican proteínas proinflamatorias. Los genes implicados en las vías del metabolismo de los lípidos también se relacionaron con el sistema inmunológico, lo que refuerza la posibilidad de una doble función clave de estos genes diferencialmente expresados. Por tanto, la mioglobina además de su papel en la calidad de la carne, parece tener una participación relevante en el metabolismo lipídico e inmunológico, e incluso un posible efecto protector contra la oxidación. Sin embargo, se deben realizar más estudios funcionales para respaldar estas evidencias.