Pfizer utiliza el desarrollo de fármacos basado en modelos para ayudar a reducir las tasas de desgaste de la fase II
“Pfizer está integrando la modelización, simulación y análisis estadístico en todo el proceso de descubrimiento y desarrollo de fármacos. Este enfoque ayuda a reducir el desgaste de la fase II al guiar la selección de la mejor vía biológica, objetivo, molécula, régimen de dosificación y población de pacientes”, explica Piet van der Graaf, director senior de Farmacometría y Farmacología Clínica de Pfizer.
Los investigadores de Pfizer utilizan software computacional como Matlab y SimBiology para apoyar el desarrollo de fármacos basado en modelos y ayudar a reducir el desgaste de la fase II. “Una base de datos cada vez mayor de estudios biológicos publicados ofrece instantáneas de los sistemas enormemente complejos que componen el cuerpo humano”, afirma el doctor Neil Benson, investigador asociado del departamento de Farmacocinética, Dinámica y Metabolismo (PDM) de Pfizer. “SimBiology nos permite construir modelos sofisticados de sistemas biológicos a partir de los datos de investigación disponibles. Mediante la simulación de estos sistemas podemos comprender mejor su cinética y dinámica y, a continuación, utilizar esa comprensión para centrar nuestra investigación en los objetivos biológicos más prometedores”.
El reto
Pfizer apuesta por el desarrollo de fármacos basado en modelos. “El desgaste de fase II debe atajarse de raíz, desde la selección de la diana biológica en las primeras fases del descubrimiento”, afirma Piet van der Graaf. “Queríamos aplicar el modelado y la simulación en las fases más tempranas de la investigación de fármacos y salvar la brecha entre la biología de sistemas y los enfoques de modelado farmacocinético-farmacodinámico (PK/PD)”.
Las interfaces basadas en texto de algunos paquetes de software de modelización pueden dificultar la construcción y puesta en común de modelos cada vez más sofisticados. “Teníamos que escribir todas las ecuaciones a mano. A medida que nuestros modelos se hacían más grandes, había que escribir muchas más ecuaciones, y construirlas y garantizar la coherencia de las unidades se hizo muy laborioso”, dice Benson. “Además, cuando necesitábamos comunicar nuestro trabajo a otros investigadores, la larga lista de ecuaciones no les resultaba fácil de entender, sobre todo si no eran expertos modelizadores”.
Los proyectos suelen requerir una importante inversión en laboratorios preclínicos para generar confianza en un planteamiento y posteriores ensayos clínicos para validar las hipótesis. Para evitar gastar millones de dólares y años de esfuerzo investigador en proyectos o enfoques que no pueden tener éxito, Pfizer quería poder identificarlos y ponerles fin antes.
La solución
Pfizer ha empezado a desarrollar un enfoque integrado de Farmacología de Sistemas en el que los investigadores utilizan herramientas como Matlab y SimBiology para modelar, simular y analizar sistemas biológicos en las primeras fases del descubrimiento y durante todo el desarrollo.
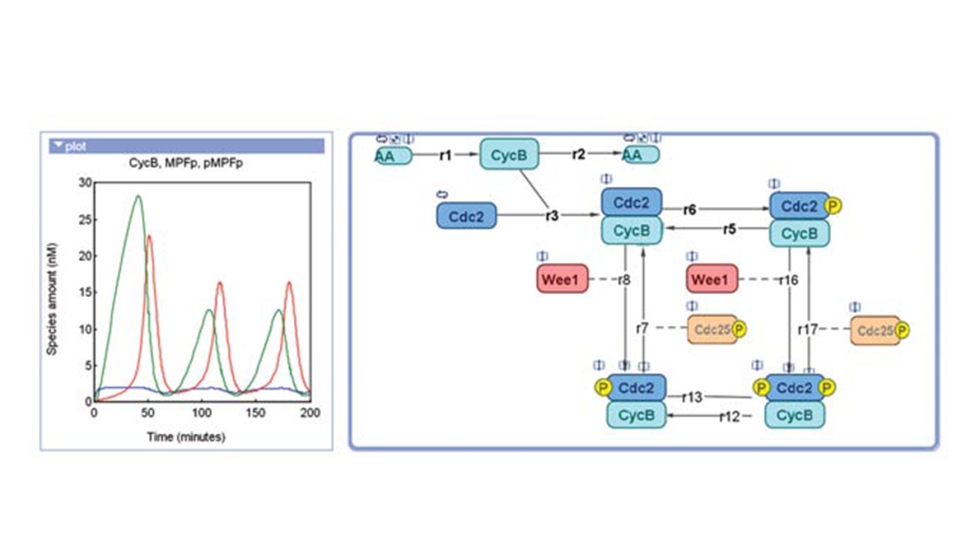
Los investigadores construyen modelos de farmacología de sistemas para responder a preguntas sobre una vía determinada, como qué receptores son probablemente los mejores objetivos y qué concentración de fármaco se necesita para lograr la inhibición requerida.
A veces, el grupo basa el modelo inicial en un modelo publicado en lenguaje de marcado de biología de sistemas (SBML), importado directamente a SimBiology y otras herramientas informáticas.
A continuación, realizan análisis de sensibilidad para identificar y clasificar los objetivos más importantes del modelo, utilizando eventos de simulación para modelizar la introducción de un fármaco concreto en el sistema.
Visualizan los resultados revisando los gráficos de diagnóstico, incluidos los gráficos de evolución temporal con y sin la presencia del fármaco.
Utilizando interfaces visuales, comparten el modelo, los gráficos y otros resultados de la simulación con el equipo del proyecto del fármaco, que proporciona estimaciones de parámetros u otras aportaciones para seguir perfeccionando el modelo.
En un proyecto, el equipo estudió la eficacia de utilizar ARN interferente de pequeño tamaño (ARNsi) para inhibir la enzima IKK en la vía NF-kB. Tras construir y simular un modelo con 26 especies y 64 parámetros, descubrieron que el nivel de inhibición que proporcionaba el ARNsi no era suficiente para lograr el resultado necesario. Los resultados experimentales posteriores concordaron con esta conclusión.
En la actualidad, Pfizer está reuniendo una biblioteca de componentes de modelos de farmacología de sistemas que pueden reutilizarse en distintos proyectos para acelerar aún más el análisis mediante modelos de PK y biología de sistemas.
Los resultados
Se profundizó en el conocimiento de la interacción fármaco-cuerpo.
“No soy matemático, pero con SimBiology he construido modelos con hasta 400 ecuaciones diferenciales ordinarias”, dice Benson. “SimBiology me permitió crear modelos más complejos de lo que había hecho nunca, y proporcionó una visión más profunda de su funcionamiento al permitir el análisis de su cinética y dinámica”.
Se evitaron costosos gastos en recursos de laboratorio y ensayos clínicos.
“En múltiples proyectos, incluido el centrado en el siRNA y la vía NF-kB, los resultados de la Farmacología de Sistemas han demostrado que un fármaco concreto no funcionaría y, en última instancia, los datos generados eran coherentes con ello”, señala Benson. “En el caso del proyecto sobre el siARN, la modelización podría haberse llevado a cabo antes de contratar ningún recurso de laboratorio”.
Aumento de la productividad de la investigación.
“Durante el descubrimiento de fármacos pueden realizarse muchos experimentos”, señala Benson. “Utilizamos los resultados de la Farmacología de Sistemas para centrar estos esfuerzos y proponer experimentos óptimos. Podemos acotar los experimentos que realizamos, centrándonos en las dianas biológicas más prometedoras”.