Software para conseguir estructuras microscópicas en tres dimensiones
18 de marzo de 2011
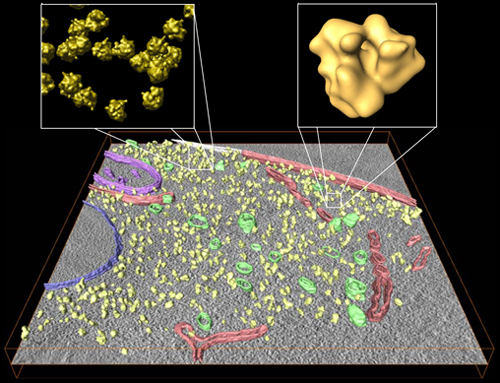
A nivel celular, la tomografía permite interpretar de forma muy acertada la función biológica de las macromoléculas que se están estudiando. Para ello se necesita conocer la estructura tridimensional, no sólo de las macromoléculas que se encuentran en el interior, sino también la de la propia célula. En el Centro Nacional de Biotecnología del CSIC por ejemplo, Cristina Risco estudia la organización en tres dimensiones de los componentes celulares que los virus manipulan dentro de la célula que han infectado. Por su parte, José Jesús Fernández trabaja en el mismo centro estudiando las diferencias en la estructura de los componentes celulares cuando se sufre alguna enfermedad neurodegenerativa.

Cómo procesar la información
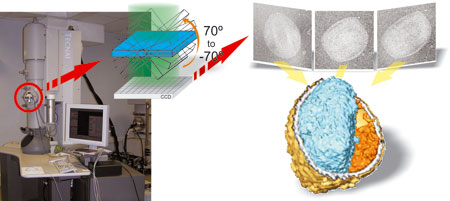
Inicialmente, para poder procesar la enorme cantidad de información obtenida, ordenando y ajustando cada una de las imágenes en la posición que ocupan una respecto a las otras, se hacía necesario el uso de multitud de ordenadores conectados en red. Sin embargo, el software disponible en la actualidad requiere tan sólo la instalación de tarjetas gráficas. Con ellas se consigue procesar los datos obtenidos en diferentes planos bidimensionales. De este modo, la tomografía nos permite construir un modelo tridimensional bastante real.
Sin embargo, la necesidad de un hardware específico hace que no todos los ordenadores puedan ser utilizados e incluso, aunque se disponga de una tarjeta gráfica compatible, a la hora llevar a cabo los procesos de cálculo requeridos, ésta debe tener unas características determinadas que la hagan funcional. Para facilitar la labor de los científicos, en el grupo de investigación de José Jesús Fernández han optimizado tanto el código de programación, como el aprovechamiento de todas las capacidades de los ordenadores estándar actuales. Combinando una técnica de computación vectorial con el uso de un procedimiento multi-thread mediante el empleo de microprocesadores multinúcleo, sin la necesidad de ningún hardware adicional, con cualquier ordenador que tenga este tipo de procesador, instrucciones SIMD y una buena memoria de caché, se puede hacer una reconstrucción tridimensional con la misma velocidad que si se hubiera instalado una tarjeta gráfica. Para ello sólo se requiere una gran cantidad de imágenes bidimensionales de los diferentes planos de la muestra a estudiar.
Un método versátil de creación de software
El método de cálculo que han desarrollado, además de alta velocidad y una resolución nanométrica, supone una gran versatilidad de cara a la creación de software para reconstruir en tres dimensiones los datos obtenidos con la tomografía por parte de cualquier otro programador. Además, al no ser precisa la instalación ni de tarjetas gráficas ni de redes de computadoras, se facilita la labor de los médicos y biólogos que van a necesitar la información de las estructuras celulares en tres dimensiones. No les hace falta la ayuda de expertos en informática para trabajar con la información estructural que les proporciona la tomografía. Tan sólo necesitan instalar un programa en sus ordenadores.
Aunque en un principio ha sido desarrollado con la idea de resolver las estructuras tridimensionales de los componentes celulares, el software es válido en cualquier situación en la que se quiera reconstruir una estructura tridimensional en poco tiempo (tomografía computarizada; tomografía electrónica; tomografía de rayos X; tomografía óptica; etc.). Por ello, puede ser utilizada tanto por biólogos o médicos, como por cualquier otro científico o ingeniero que trabaje en el estudio de materiales.























