En la actualidad se carece de estrategias y medios eficaces para el control de la enfermedad
Identificación in-situ de viñas afectadas por yesca mediante imagen hiperespectral
Ainara López Maestresalas, Jon Ruiz de Gauna, Carmen Jarén y Silvia Arazuri
Departamento de Ingeniería, Universidad Pública de Navarra, Campus de Arrosadia, 31006, Pamplona
En este estudio se demuestra cómo el uso combinado de un sensor de imágenes hiperespectrales in-situ, con el análisis discriminante por mínimos cuadrados parciales, permite identificar hojas de vid afectadas por la enfermedad de la yesca.
Introducción
La yesca es una de las enfermedades fúngicas de la madera de la vid que representa una de las mayores amenazas para la viticultura moderna, ya que causa grandes pérdidas económicas anuales (Ouadi et al., 2019). Estas pérdidas se deben principalmente a la reducción del rendimiento y la calidad de la uva y, en algunas ocasiones, a la muerte de la vid en un periodo de tiempo variable (Fontaine et al., 2016).
La yesca no es una enfermedad única, sino un grupo de síndromes causados por una serie de hongos ascomicetos y basidiomicetos. Las principales especies implicadas son Phaeomoniella chlamydospora (Gams y Crous, 2000), y Phaeoacremonium aleophilum en los ascomicetos, mientras que Fomitiporia mediterranea es el principal patógeno basidiomiceto (Fischer, 2002). Estas especies infectan la planta mediante esporas, que pueden ser transportadas por el aire o la lluvia y entrar en la planta a través de heridas en la madera causadas por impactos o por trabajos mecánicos como podas, etc. (Bertsch et al., 2013).

En la actualidad se carece de estrategias y medios eficaces para el control de la enfermedad. El único producto químico conocido capaz de controlar la enfermedad es el arsenito sódico y su uso se prohibió en 2003 (Fontaine et al., 2016). Las técnicas aprobadas para controlar esta enfermedad incluyen métodos preventivos como el uso de fungicidas en las heridas de las plantas, el uso de sistemas de plantación que no requieran podas intensivas o la adquisición de material vegetal sano en viveros (Fontaine et al., 2016; Mondello et al., 2018).
Por ello, es aconsejable monitorizar el cultivo para identificar aquellas vides susceptibles a la enfermedad. En este sentido, sensores ópticos como las imágenes hiperespectrales (HSI) se presentan como herramientas prometedoras para la evaluación no invasiva del estado fisiológico de las plantas y para el diagnóstico y detección de enfermedades (Mahlein, 2016; Zhang et al., 2019). Así, la técnica HSI combinada con un enfoque quimiométrico puede ser muy útil para la detección y cuantificación de la enfermedad (Bock et al., 2010), en estadios de desarrollo tempranos cuando todavía los síntomas no son apreciables en la hoja.
Por este motivo, en este estudio se utilizó una cámara hiperespectral para la detección in-situ de la enfermedad de la yesca en hojas de vid del cultivar Tempranillo.
Materiales y métodos

Las imágenes se capturaron in-situ en condiciones de luz ambiental natural el 21 de agosto de 2023. Se adquirió una imagen por vid, es decir, se obtuvieron 11 imágenes individuales.

Una vez obtenidas las imágenes, se dividieron aleatoriamente en un conjunto de calibración, formado por una imagen de una vid asintomática (clase 1) y ocho vides sintomáticas (clase 2), y un conjunto de validación, formado por una imagen de cada clase. Los grupos de calibración y validación se procesaron de forma diferente. Las imágenes del grupo de calibración se utilizaron para seleccionar aleatoriamente los píxeles que formarían la matriz de calibración; se seleccionaron 100 píxeles de la imagen de la clase 1 y 100 de las imágenes de la clase 2. Esta selección se llevó a cabo con el software HYPER-Tools 3.0, que funciona en el entorno MATLAB R2023a. El resultado fue una matriz bidimensional con 200 filas y 204 columnas correspondientes a las longitudes de onda.
Por otro lado, las 2 imágenes que conformaban el grupo de validación se segmentaron individualmente para separar la región de interés (ROI) del fondo. Para ello, se utilizó el algoritmo k-means implementado en MATLAB. A continuación, se realizó el despliegue, que consistió en transformar la información almacenada en la imagen original (3 dimensiones) en una matriz bidimensional (2D) con la reflectancia de cada píxel en cada longitud de onda. Se obtuvieron así dos matrices independientes de 120209 filas y 204 columnas y de 122348 filas y 204 columnas para las vides asintomáticas y sintomáticas, respectivamente.
A continuación, se empleó el análisis discriminante por mínimos cuadrados parciales (PLS-DA) para clasificar las muestras en las clases 1 (asintomáticas) y 2 (sintomáticas). El análisis PLS-DA es una técnica de reconocimiento de patrones que utiliza la regresión PLS para predecir clases o grupos de asignación a partir del espectro de las muestras. Para utilizar este método, se crea una matriz binaria (Y) con el mismo número de filas que muestras y el mismo número de columnas que clases. De este modo, PLS-DA realiza una calibración cualitativa, es decir, en lugar de calibrar para una variable continua, calibra para la pertenencia a una clase (Shenk y Westerhaus, 1995).
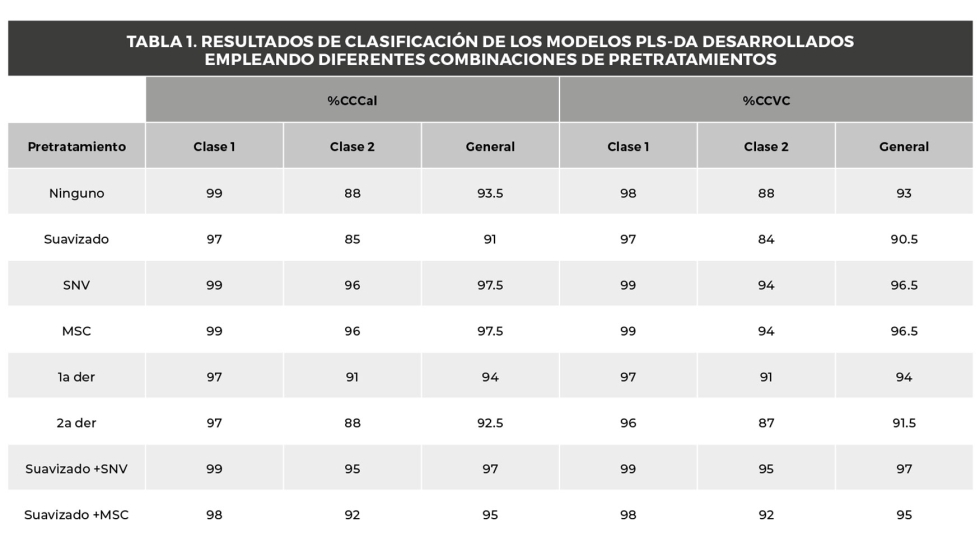
Además, se aplicaron diferentes pretratamientos matemáticos a los espectros, como el suavizado, la primera (1ª der) y la segunda derivada (2ª der) de Savitzky Golay (SG) (con polinomio de segundo orden y 15 puntos de ventana), la Variable Normalizada Estándar (SNV) y la Corrección Multiplicativa de la Dispersión (MSC), de forma individual o combinados. También se analizó el efecto de la ausencia de pretratamiento (Ninguno). Se llevó a cabo una validación cruzada (VC) mediante persianas venecianas con 10 divisiones.
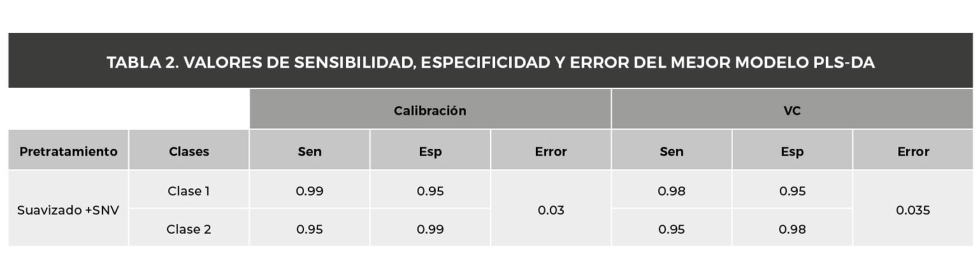
Para evaluar la eficacia de los modelos, se tuvieron en cuenta principalmente el porcentaje de muestras correctamente clasificadas (%CC) (precisión) en cada clase, así como la sensibilidad, la especificidad y el error de clase en los conjuntos de datos de calibración y VC. El modelo de mejor rendimiento se utilizó para la predicción externa de las muestras. Por último, se representaron imágenes de clasificación para visualizar la distribución de los píxeles estimados como clase 1 o 2 utilizando código propio.
Se utilizó el programa informático PLS_Toolbox v.9 en MATLAB R2023a para aplicar los distintos pretratamientos y construir los modelos PLS-DA.
Resultados y discusión
La Figura 3 (b, d) muestra las imágenes de clasificación obtenidas utilizando el modelo PLS-DA con la combinación de Suavizado + SNV, con sus correspondientes fotografías (Figura 3 (a, c)). En la Figura 3c se observan algunos síntomas foliares de yesca. En la clase 1 (asintomática) un 94.46% de los píxeles se clasificaron correctamente como asintomáticos (píxeles de la Figura 3b en azul), mientras que para los píxeles sintomáticos se realizó una predicción a ciegas (Figura 3d).
La Figura 3b muestra algunos casos de falsos positivos, indicados por los píxeles de color naranja, es decir, píxeles asintomáticos (sanos) clasificados como sintomáticos (con yesca). Estos se observan en el extremo superior de la imagen, correspondiente a los pecíolos de las hojas, y en las propias hojas, correspondientes a otros síntomas que no son yesca. La Figura 3d muestra como la mayoría de los píxeles clasificados como sintomáticos (píxeles en naranja) eran, de hecho, sintomáticos, como puede observarse en la Figura 3c.
Los resultados obtenidos indican la viabilidad de identificar las vides que presentan síntomas de infección por yesca en una fase temprana, cuando los síntomas son apenas perceptibles. Esta información permitiría una gestión diferenciada de las vides identificadas como afectadas por yesca, además de facilitar el seguimiento anual de la incidencia de la enfermedad en el viñedo.

Conclusiones
El modelo PLS-DA desarrollado aplicando la combinación de pretratamientos suavizado seguido de SNV obtuvo los mejores resultados, con una tasa de precisión en la validación cruzada del 97%. Además, las imágenes de clasificación facilitaron la visualización de la distribución espacial de los píxeles asintomáticos y sintomáticos.
A pesar de las limitaciones de este estudio, incluido el bajo número de muestras utilizadas, entre otras, puede concluirse que la tecnología HSI tiene potencial para la detección in-situ de la yesca y posiblemente de otras enfermedades de la madera de la vid.
Referencias
Bertsch, C., Ramírez-Suero, M., Magnin-Robert, M., Larignon, P., Chong, J., Abou-Mansour, E., Spagnolo, A., Clément, C., Fontaine, F., 2013. Grapevine trunk diseases: Complex and still poorly understood. Plant Pathology, 62 (2), 243-265. https://doi.org/10.1111/j.1365-3059.2012.02674.x
Bock, C. H., Poole, G. H., Parker, P. E., Gottwald, T. R., 2010. Plant disease severity estimated visually, by digital photography and image analysis, and by hyperspectral imaging. Critical Reviews in Plant Sciences, 29 (2), 59-107. https://doi.org/10.1080/07352681003617285
Fischer, M., 2002. A new wood-decaying basidiomycete species associated with esca of grapevine: Fomitiporia mediterranea (Hymenochaetales). Mycological Progress, 1 (3), 315-324. https://doi.org/10.1007/s11557-006-0029-4
Fontaine, F., Gramaje, D., Armengol, J., Smart, R., Nagy, Z. A., Borgo, M., Rego, C., Corio-Costet, M.-F., 2016. Grapevine trunk diseases. A review (p. 24). OIV Publications.
Gams, W., & Crous, P. W. (2000). «Phaeomoniella chlamydospora» Gen. Et Comb. Nov., a Causal Organism of Petri Grapevine Decline and Esca. Phytopathologia Mediterranea, 39 (1), 112-118. https://doi.org/10.1400/57828
Mahlein, A.-K., 2016. Plant disease detection by imaging sensors—Parallels and specific demands for precision agriculture and plant phenotyping. Plant disease, 100 (2), 241-251. https://doi.org/10.1094/PDIS-03-15-0340-FE
Mondello, V., Songy, A., Battiston, E., Pinto, C., Coppin, C., Trotel-Aziz, P., Clément, C., Mugnai, L., Fontaine, F., 2018. Grapevine trunk diseases: A review of fifteen years of trials for their control with chemicals and biocontrol agents. Plant Disease, 102 (7), 1189-1217. https://doi.org/10.1094/PDIS-08-17-1181-FE
Ouadi, L., Bruez, E., Bastien, S., Vallance, J., Lecomte, P., Domec, J.-C., Rey, P., 2019. Ecophysiological impacts of Esca, a devastating grapevine trunk disease, on Vitis vinifera L. PLOS ONE, 14 (9), e0222586. https://doi.org/10.1371/journal.pone.0222586
Shenk, J. S., Westerhaus, M. O., 1995. Routine operation, calibration, development and network system management manual. NIRSystems Inc., Silver Spring, MD, USA.
Zhang, J., Huang, Y., Pu, R., Gonzalez-Moreno, P., Yuan, L., Wu, K., Huang, W., 2019. Monitoring plant diseases and pests through remote sensing technology: A review. Computers and Electronics in Agriculture, 165, 104943. https://doi.org/10.1016/j.compag.2019.104943
Los resultados obtenidos indican la viabilidad de identificar las vides que presentan síntomas de infección por yesca en una fase temprana, cuando los síntomas son apenas perceptibles

























