La mejora genética de judía podría incorporar métodos convencionales y otros surgidos del conocimiento del genoma de esta leguminosa
La judía común (Phaseolus vulgaris L.): una planta modelo para estudios genéticos en leguminosas
La judía común (Phaseolus vulgaris L.) representa en la actualidad una de las leguminosas más importantes para consumo humano desde el punto de vista nutricional debido a su aporte de proteínas, hidratos de carbono, elementos esenciales y vitaminas (Broughton et al. 2003). Representa el 50% de las leguminosas de grano que se consumen en todo el mundo, y su producción es casi el doble que la de garbanzos, el segundo cultivo de leguminosas grano más consumido como alimento. La judía común se cultiva por sus hojas verdes, vainas verdes y semillas inmaduras y/o secas.

1. Importancia y organización actual del cultivo de judía común
1.1. Importancia económica y social de la judía común
En los países en desarrollo, la judía común (Phaseolus vulgaris) es considerada como la principal fuente de proteínas debido al acceso limitado a la proteína animal. En este sentido, y desde una perspectiva nutricional, la judía común constituye el 40%, 31% y 15% de la ingesta diaria de proteínas totales en países como Burundi, Ruanda y Uganda, respectivamente. E incluso para un productor importante como Brasil, la judía común proporciona el 9% de la proteína en la dieta.
La producción anual de judía para grano seco es aproximadamente 15 millones de toneladas, siendo el rendimiento medio de 700 kg ha-1; no obstante, los rendimientos en algunos países pueden alcanzar los 3.000 kg ha-1. Los mayores productores de judía seca son Brasil, México, China y EE UU. Por su parte, la producción anual de judía verde es aproximadamente 4,5 millones de toneladas, con la mayor producción en el Mediterráneo y en EE UU (FAOSTAT 2010; http://faostat3.fao.org).
La importancia de la judía común como leguminosa de grano para la dieta humana, junto a su papel como un cultivo social, han hecho que su cultivo y mejora varietal sea una preocupación creciente para los organismos internacionales. Es por ello que hace unos años se creó el consorcio internacional 'PhasIbeam' para la secuenciación del genoma y transcriptoma de la judía común (www.cyted.org/es), cuyo objetivo primordial no era otro que, a partir del conocimiento de los genes responsables de las características agronómicas importantes para el cultivo, poder contribuir al desarrollo de nuevas variedades mejor adaptadas, más productivas y sostenibles, tanto para los agricultores como los consumidores finales.
1.2. Estructura poblacional de la judía común
Los estudios basados en la variación genética de la proteína faseolina, esencial durante el almacenamiento de la semilla (Gepts et al. 1986; Gepts 1990), de caracteres morfológicos de la planta (Figura 1A; Gepts y Debouck 1991),
junto con la aplicación de marcadores moleculares (Becerra Velázquez y Gepts 1994; Koenig y Gepts 1989; Tohme et al. 1996; McClean et al. 2004), han permitido establecer que la judía común ha sido domesticada al menos dos veces (Getps et al. 1986; Koenig y Gepts 1989; Bitocchi et al. 2012). Tales procesos de domesticación reflejan la distribución geográfica de los progenitores silvestres, en Mesoamérica, en una región que abarca México, Centroamérica, Colombia y Venezuela, y en la región sur de los Andes, que abarcar el sur de Perú, Bolivia y Argentina (Figura 1B). Ambos orígenes de domesticación han dado lugar a dos acervos genéticos diferenciados que convergen en Colombia (Gepts et al. 1986). Además, estudios posteriores sugieren la existencia de un tercer centro de origen de la especie, denominado el Norte Andino, situado en Perú y Ecuador (Islam et al. 2002; Debouck et al. 1993). Después de la domesticación, esta divergencia genética propició la aparición de tres razas dentro del acervo genético Andino, Nueva Granada, Perú y Chile (Singh et al. 1991), y cuatro razas dentro del acervo Mesoamericano, Durango, Jalisco, Mesoamérica, y Guatemala (Singh et al. 1991; Beebe et al. 2000). Estos eventos de domesticación independiente contrastan con lo acontecido con especies como el maíz (Matsuoka et al. 2002) y la soja (Glycine max; Powell et al. 1996; Xu y Gai 2003), donde una población; domesticada surgió de una sola subespecie o especie relacionada, respectivamente.
El conocimiento del origen diferente de los progenitores silvestres y domesticados de la judía común, así como de las relaciones genéticas entre ellos, han permitido conocer algunos de los genes implicados en el proceso de domesticación, y por ende, responsables de las diferencias morfológicas o fisiológicas entre ambos acervos genéticos. A modo de ejemplo, las variaciones alélicas en el gen TERMINAL FLOWER 1 (TFL1) de judía (Kwak et al. 2008; Repinski et al. 2012; Figura 1C), han posibilitado la aparición de cultivares de crecimiento determinado (tallo principal y laterales que terminan en una inflorescencia terminal), a partir de genotipos indeterminados (crecen indefinidamente produciendo tejidos vegetativos –tallos y hojas– y reproductivos –flores y frutos–), lo que a su vez ha influido de manera decisiva en la diversificación del cultivo de judía, en el que actualmente se cuenta con variedades determinadas y erectas, idóneas para producción de grano, y variedades indeterminadas, adecuadas para cultivo intensivo y producción de vaina fresca.
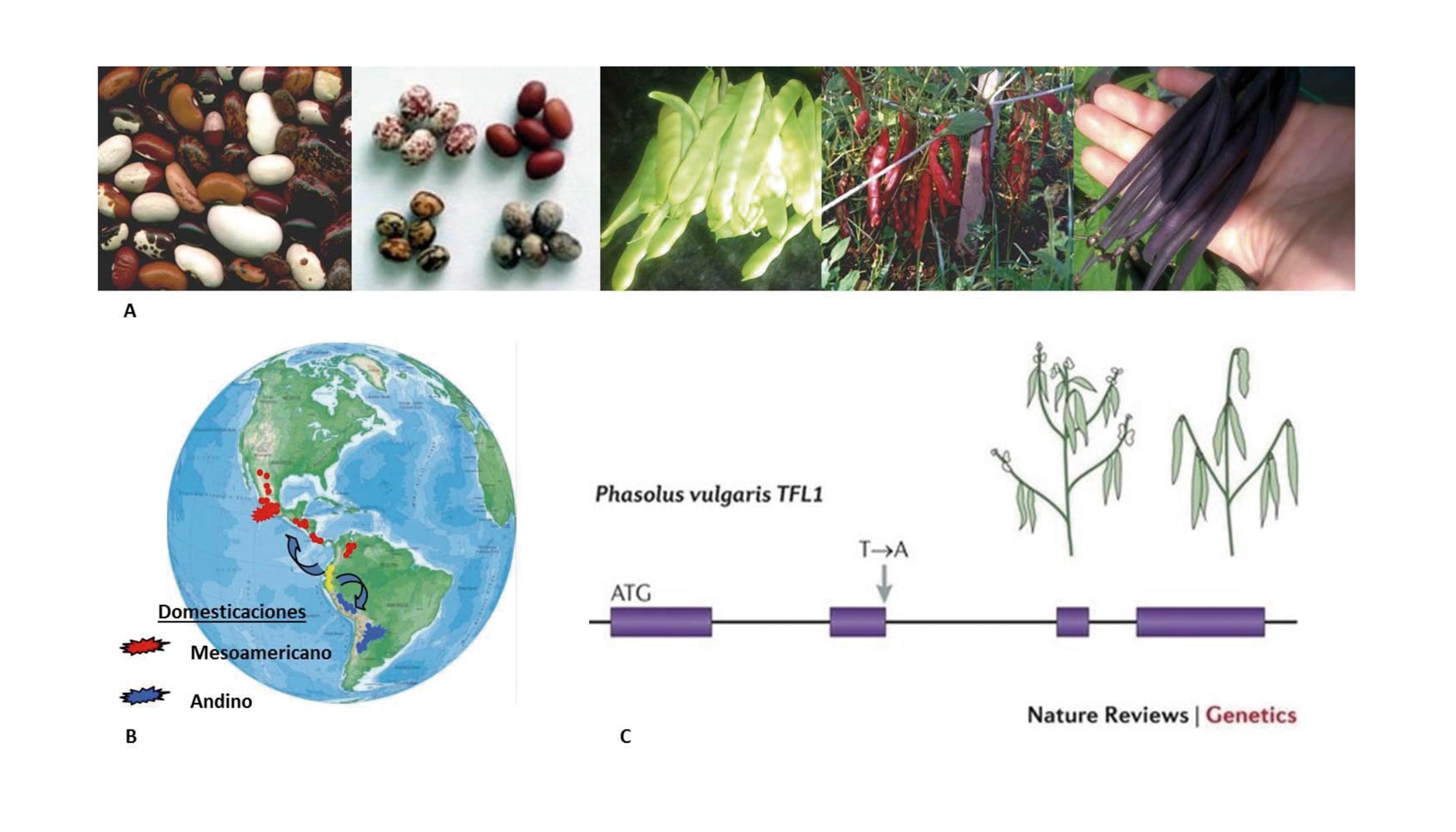
Figura 1. A) Variabilidad del fruto, vainas y semillas, en judía común. B) Los dos acervos genéticos geográficos de judía común, denominados Mesoamericano (rojo) y Andino (azul). C) Variación genética en el gen TFL1 de judía (cambio de un nucleótido T -> A) que ha influido de manera decisiva en el proceso de domesticación de judía común promoviendo la aparición de cultivares determinados (figura adaptada de Nature Reviews Genetics).
1.3. Relaciones filogenéticas de la judía común con otras especies de leguminosas
Los principales cultivos de leguminosas se dividen en dos clados (Figura 2A), dentro del grupo de papilionáceas, galegoide y phaseoloide (Cronk et al. 2006). En general, las especies del clado galegoide (por ejemplo, guisante, lenteja, garbanzo, haba) florecen en condiciones de día largo y habitan en regiones templadas, mientras que las del clado phaseoloide (por ejemplo, soja, garbanzo, y judía común), por lo general se localizan en latitudes más bajas y suelen ser especies de día corto (Summerfield y Roberts, 1985). Otras dos leguminosas importantes, cacahuete (Arachis hypogaea) y altramuz (Lupinus spp.), tienen una posición basal a estos dos clados, y con orígenes en latitudes bajas y altas, respectivamente.
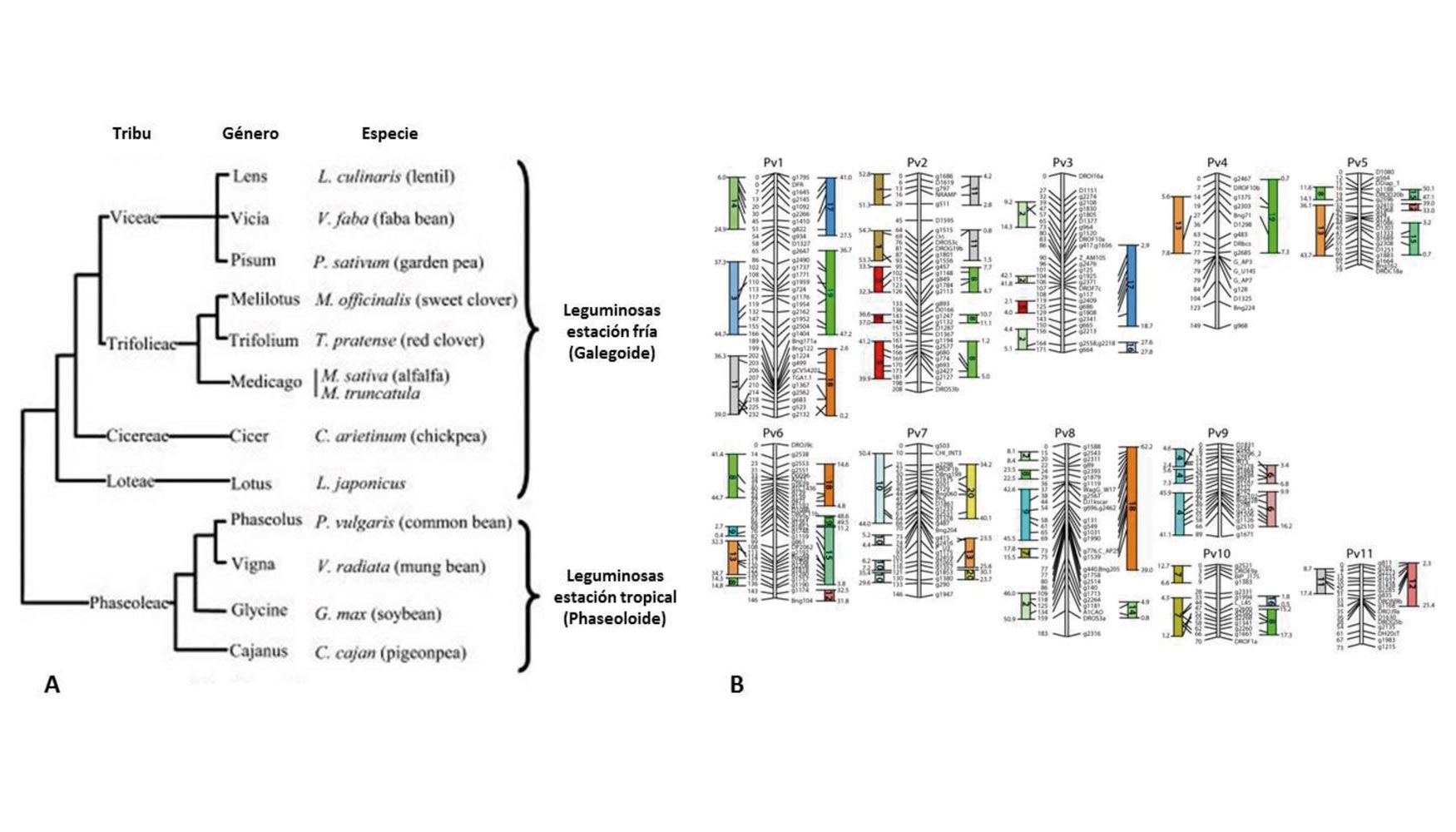
Figura 2. A) Relaciones filogenéticas de las leguminosas Papilonoideas. B) Relaciones de sintenía entre los genomas de judía común (marcadores situados en los distintos grupos de ligamiento Pv) y soja (rectángulos coloreados). Figura adaptada de McClean et al. (2010).
A pesar de que la soja y la judía común son especies estrechamente relacionadas, ambas líneas evolutivas se separaron hace aproximadamente 15 millones de años (Lavin et al. 2005). Además, en el género Glycine se han producido duplicaciones del genoma seguidas por diferentes tipos de reordenamiento cromosómicos que, en suma, han conducido a la diploidización del genoma cuyo número cromosómico actual es 2n = 40. Los análisis de sintenia indican que la mayoría de los segmentos de un grupo de ligamiento de la judía común (Pv en la Figura 2B) son muy similares a los dos cromosomas de soja (Galeano et al. 2009). De hecho, McClean et al. (2010) han comprobado con éxito la hipótesis de que el genoma de judía común es una versión diploide del genoma de la soja, para lo cual han comparado la posición y ordenación de los genes mapeados de judía común (McConnell et al. 2010) en los 20 cromosomas del genoma de soja (Figura 2B).
En este marco evolutivo, la judía común ha demostrado ser útil como modelo para entender la organización de un genoma de mayor tamaño y complejidad como es el de la soja (alrededor de 1100 Mbp), pero también para realizar estudios de genómica comparada con otras leguminosas como Medicago truncatula y Lotus japonicus. A buen seguro, ello permitirá identificar nuevos genes y funciones génicas relevantes para muchos de los caracteres de importancia agronómica (arquitectura de planta, resistencia a enfermedades y plagas, adaptación a factores de estrés ambiental, producción y calidad de frutos y semillas). De esta manera, Quiroga et al. (2015a) identificaron diez miembros de la familia FT (FLOWERING LOCUS T)/TFL1 en el genoma de judía común (Figura 3), y pusieron de manifiesto que la expansión de la familia FT/TFL1 ocurrió relativamente temprano en la evolución de las leguminosas (Figura 3), siendo ambos genes, factores esenciales que controlan el proceso de floración dependiendo de condiciones de fotoperiodo.
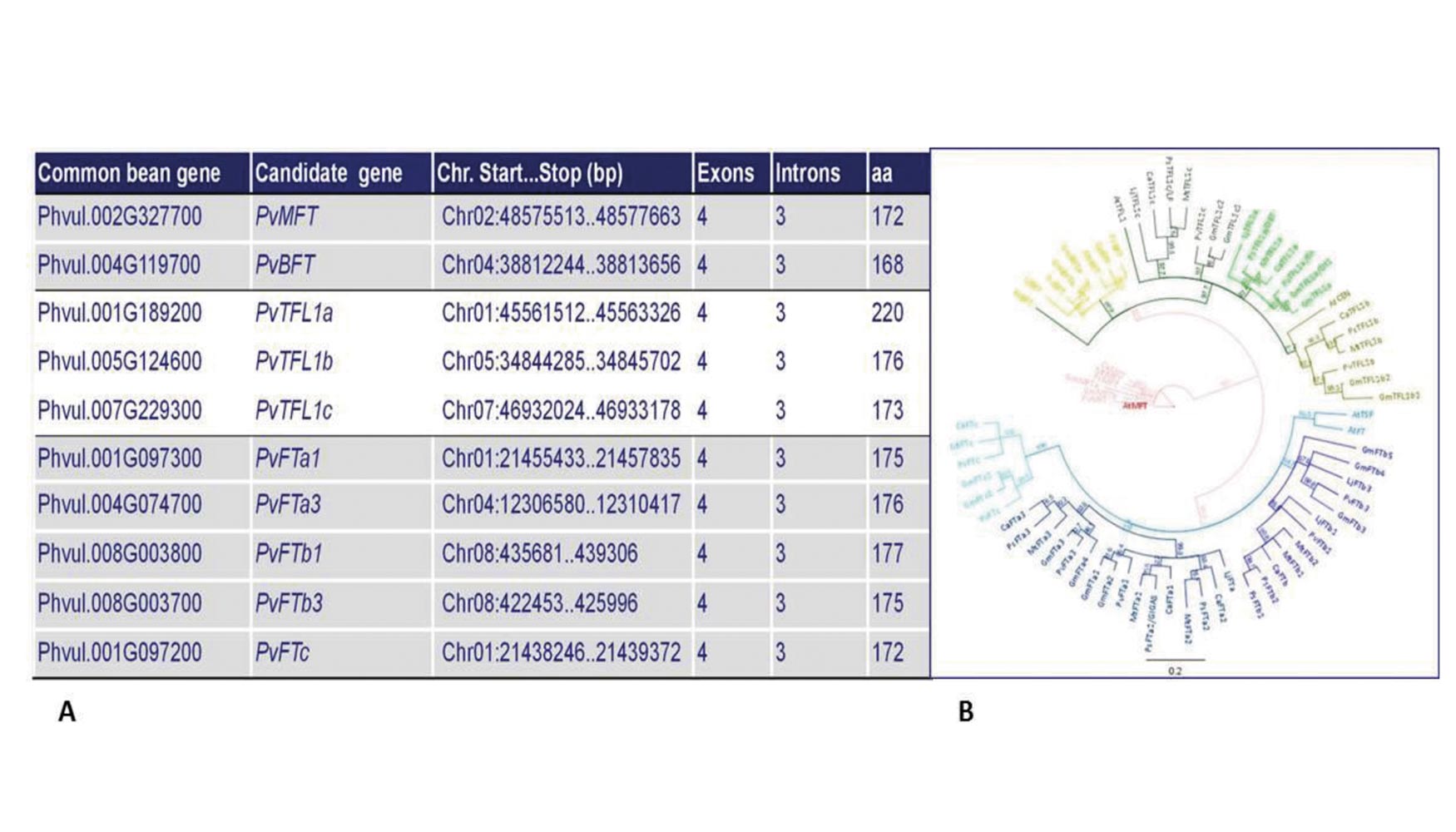
Figura 3. Familia de genes FT/TFL1 en judía común (izquierda). Filogenia de los homólogos FT/TFL1 de Arabidopsis y otras especies de leguminosas (derecha). At = Arabidopsis thaliana, Mt = Medicago trunculata, Gm = Glycine max, Ca = Cicer arietinum, LJ = Lotus japonicus, Ps = Pisum sativum y Pv = Phaseolus vulgaris.
Estos recursos genómicos adicionales, especialmente el mapeo genético y físico de genes y marcadores de ADN, proporcionan herramientas genéticas que no solo mejoran nuestros conocimientos sobre el genoma de judía y otras leguminosas, sino que se pueden utilizar de manera inmediata como criterio inequívoco para una selección eficaz y rápida de líneas (cultivares y variedades) de interés agronómico.
2. Marcadores y mapas genéticos en judía común
2.1 Limitaciones de la mejora genética y ventajas de la selección asistida con marcadores moleculares
Los procedimientos tradicionales para la mejora genética de judía común, que se basan en la selección fenotípica y los ensayos de progenie, han sido muy eficaces en la mejora de la productividad y calidad de este cultivo durante las últimas décadas (Duc et al. 2015; Kelly 2010; Santalla et al. 2005). Estos métodos, sin embargo, son laboriosos, requieren de periodos de ejecución prolongados, y conllevan algunas dificultades experimentales. De hecho, la duración media de un proyecto de mejora genética de judía utilizando exclusivamente métodos convencionales de hibridación y selección, suele ser de 10 a 15 años hasta la introducción en el mercado de una nueva variedad. A menudo, este proceso resulta excesivamente largo para satisfacer las demandas de aumento de productividad y calidad por parte de productores y consumidores. Más aún, algunas características agronómicas importantes, entre ellas la resistencia a determinadas enfermedades y plagas, la tolerancia a factores de estrés abiótico y la calidad nutricional del fruto, sólo se pueden encontrar en especies o formas silvestres (Porch et al. 2013), siendo la introgresión de esta variabilidad genética en las variedades cultivadas a menudo una dificultad añadida al proceso de mejora toda vez que se precisan de ciclos de retrocruzamiento y autofecundación prolongados en el tiempo. Además, durante este proceso, algunos de los genes de interés del donante silvestre se pueden perder, lo que limita el nivel de expresión del carácter en la progenie. Por otra parte, es preciso eliminar la gran mayoría de los genes no deseados introducidos desde el donante silvestre (lastre genético) para que la nueva variedad no arrastre características que mermen su producción o calidad comercial. Estos y otros problemas asociados con el uso de métodos convencionales de mejora del cultivo justifican el empleo de técnicas más eficientes.
Un enfoque alternativo pero complementario, para mejorar la eficiencia de la selección es a través de marcadores genéticos asociados, a través de ligamiento o pleiotropia, con genes o QTLs (Quantitative Trait Loci) que controlan caracteres de interés agronómico. Además, el uso de marcadores y mapas genéticos facilita la determinación del número, localización cromosómica, y los efectos individuales e interacciones de genes o QTL que afectan a características deseables. Tras su identificación, los genes o QTL útiles se pueden introducir en el material vegetal adecuado a través de selección asistida por marcadores (Marker Assisted Selection, MAS). MAS no sólo puede acelerar el proceso de transferencia de genes, sino también puede permitir incorporar (piramidar) varios genes y QTL favorables a partir de diferentes donadores, lo que supone un método complementario eficaz para la mejora de la judía común. De hecho, en los últimos años y gracias a la aplicación de MAS, los trabajos de mejora genética en judía común, y en otras leguminosas, han tenido un notable progreso (Miklas et al. 2006)
2.2 Mapas genéticos y mapeo de genes y/o QTLs en judía común
En la actualidad, se encuentran disponibles más de 25 mapas genéticos para judía común que incluyen diferentes caracteres y tipos de marcadores moleculares. Estos se han obtenido a partir de poblaciones segregantes en los dos acervos genéticos (Kelly et al. 2003; Yuste et al. 2012; González et al. 2015). De igual manera, se ha establecido un mapa consenso de referencia en judía común, que permite correlacionar los resultados de diferentes análisis de ligamiento y trabajos de cartografía genética, con grupos de ligamiento previamente asignados (Freyre et al. 1998).
Recientemente, otros nuevos tipos de marcadores genéticos se están empleando para el mapeo genético en judía común como TRAP (Targeted Region Amplification Polymorhism) y RGA (Resistance-Gene Analogs)-marcadores de genes de resistencia a enfermedades (Miklas et al. 2006). También, el mapeo con marcadores SNP (Single Nucleotide Polymorphism) y de secuencias de EST (Expressed Sequence Tags) está permitiendo el desarrollo de mapas funcionales en judía común que facilitan el establecimiento de correlaciones entre genes candidatos y QTLs específicos. En cualquier caso, los mapas genéticos de judía común basados en marcadores moleculares se han complementado en los últimos años con mapas físicos construidos a partir de la secuenciación de cromosomas bacterianos artificiales (Bacterial Artificial Chromosome, BAC), y sobre todo, a partir de la información obtenida de los proyectos de secuenciación del genoma de esta especie.
3. El genoma de judía común
3.1. Secuenciación del genoma
La secuencia completa del genoma de judía común se ha obtenido a partir de dos líneas, cada una perteneciente a un acervo genético diferente, a saber, BAT93, una línea mesoamericana, y G19883, de origen andino. Los genomas de BAT93 y G19883 tienen un tamaño de 550 Mbp y 473 Mbp, y organizados en 11 cromosomas. En dichos genomas se han anotado 30.491 y 27.197 genes, respectivamente, que codifican proteínas hipotéticas. Los resultados detallados de ambos proyectos de secuenciación fueron publicados por un grupo de Estados Unidos (McClean et al. 2013; http://www.phytozome.net/commonbean.php), en el caso de G19883, y por el consorcio PhasIbeam antes mencionado, integrado por grupos de España, Brasil, Argentina y México (http://mazorka.langebio.cinvestav.mx/Phaseolus/), en el caso de BAT93 (Schmutz et al. 2014; Vlasova et al. 2016). La comparación del genoma de ambas líneas de judía indica que de los 25.991 loci de BAT93 que se localizan en cromosomas, 20.617 fueron mapeados de forma única en el genoma andino.
3.2. Tecnologías emergentes: TILLING y transformación genética
La herramienta TILLING (Targeted Induce Local Lesions In Genomes) permite identificar nuevas variantes genéticas de genes específicos, a partir de una colección de mutantes inducidos artificialmente. En judía común, el grupo BAS-MBG está desarrollando una plataforma de TILLING (Figura 4; Quiroga et al. 2015b) a partir de más de 5.500 familias M2 obtenidas en un programa de mutagénesis con EMS (Ethyl Methanesulfonate). Esta plataforma de TILLING se beneficiará de los resultados de secuenciación del genoma de judía común y otras especies de leguminosas, como la soja, proporcionando nuevas líneas de judía de utilización directa e inmediata en programas de mejora genética.
Por otra parte, se sabe que la judía común es una especie recalcitrante a la transformación genética. Sin embargo, recientemente, Estrada-Navarrete et al. (2006) modificaron un protocolo desarrollado originalmente en la soja para la transformación con Agrobacterium rhizogenes en judía común. Esta disponibilidad de raíces transgénicas proporcionará un nuevo método para la sobre-expresión o supresión de genes endógenos, especialmente los que participan en los caracteres agronómicos dependientes de la raíz, lo que a su vez permitirá conocer la función de dichos genes y diseñar estrategias de mejora genética convencional.
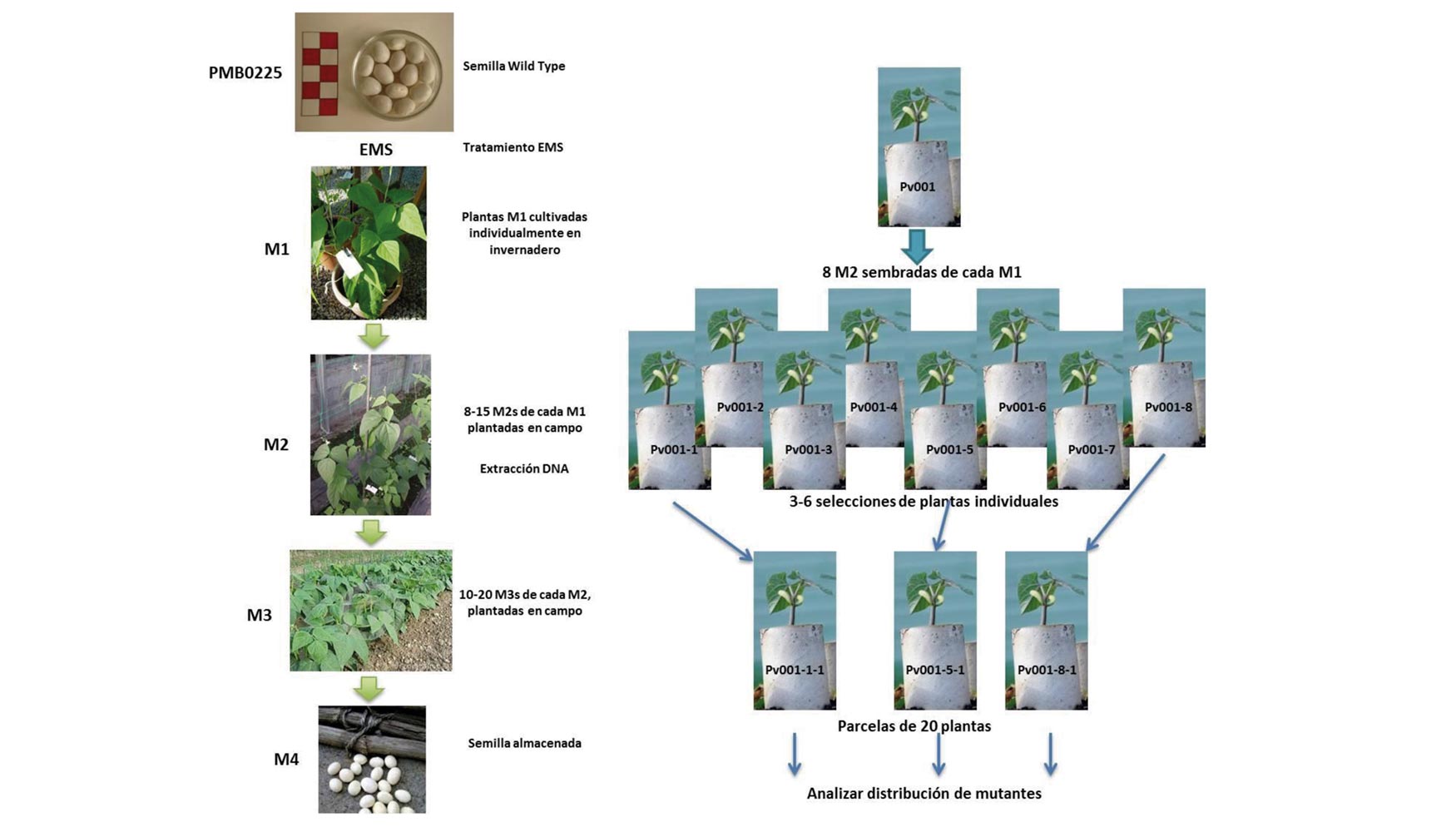
Estas nuevas herramientas surgidas del conocimiento del genoma se vienen a sumar a otras características genéticas interesantes desde el punto de vista agronómico, entre ellas, el ser una especie diploide (2n = 22), con un genoma relativamente pequeño (587 Mbp), y la disponibilidad de una amplia gama de variación genética tanto de formas silvestres como cultivadas existente en los bancos de germoplasma. Todas ellas, unidas a la facilidad de cultivo en una amplia gama de ambientes, un ciclo de crecimiento corto, la insensibilidad al fotoperiodo, altas tasas de fecundidad y homozigosis y un gran potencial reproductivo y su naturaleza estrictamente autógama, convierten a la judía común en un excelente sistema modelo tanto para investigación básica como aplicada (Santalla et al. 2002).
Agradecimientos
Este trabajo fue parcialmente financiado por el Ministerio de Economía y Competitividad (AGL2014-51809, RF2012-0026-C02-01 y RF2012-00026-C02-02), el programa UE-FEDER, y la Junta de Andalucía (P12- AGR–01482). Los autores agradecen al Campus de Excelencia Internacional Agroalimentario - CeiA3 el apoyo en actividades de I+D.
Referencias
- Beebe S, Rengifo J, Gaitán E, Duque MC, Tohme J. 2001. Diversity and origin of Andean landraces of common bean. Crop Sci. 41:854–862.
- Becerra Velázquez VL, Gepts P. 1994. RFLP diversity in common bean (Phaseolus vulgaris L.). Genome 37: 256-263.
- Bitocchi E, Nannia L, Belluccia E, Rossia M, Giardinia A, Spagnoletti Zeulib P, Logozzob G, Stougaardc J, McCleand P, Attenee G, Papa R. 2012. Mesoamerican origin of the common bean (Phaseolus vulgaris L.) is revealed by sequence data. Proc. Natl. Acad. Sci. USA 109: 14.
- Broughton WJ, Hernández G, Blair M, Beebe S, Gepts P, Vanderleyden J. 2006. Beans (Phaseolus spp.) – model food legumes. Plant and Soil 252 (1): 55-128.
- Cronk Q, Ojeda I, Pennington RT. 2006. Legume comparative genomics: progress in phylogenetics and phylogenomics. Curr Opin Plant Biol 9: 99–10.
- Debouck DG, Toro O, Paredes OM, Johnson WC, Gepts P. 1993. Genetic diversity and ecological distribution of Phaseolus vulgaris in northwestern South America. Econ. Bot. 47: 408–423.
- Duc G, Agrama H, Bao S, Berger J, Bourion V, Burstin J, Burton J, De Ron AM, Gowda CL, Mikic A, Millot D, Singh K, Tullu A, Vandenberg A, Vaz Patto MC, Warkentin T, Zong X. 2015. Breeding annual legumes for adaptation to low imput cropping systems and new areas: methods to approach more complex traits and target new variety ideotypes. Critical Reviews in Plant Sciences 34 (1-3): 381-411.
- Estrada-Navarrete G, Alvarado-Affantranger X, Olivares JE, Díaz-Camino C, Santana O, Murillo E, Guillén G, Sánchez-Guevara N, Acosta J, Quinto C, Li D, Gresshoff PM, Sánchez F. 2006. Agrobacterium rhizogenes transformation of the Phaseolus spp.: A tool for functional genomics. Mol. Plant Microbe Interact. 19: 1385–1393.
- Freyre R, Skroch PW, Geffroy V, Adam-Blondon AF, Shirmohamadali A, Johnson WC, Llaca V, Nodari RO, Pereira PA, Tsai SM, Tohme J, Dron M, Nienhuis J, Vallejos CE, GeptsP. 1998. Towards an integrated linkage map of common bean. 4. Development of a core linkage map and alignment of RFLP maps. Theor. Appl. Genet. 97: 847–856.
- Galeano CH, Fernández AC, Gómez M, Blair MW. 2009. Single strand conformation polymorphism based SNP and Indel markers for genetic mapping and synteny analysis of common bean (Phaseolus vulgaris L.). BMC Genomics 10: 629.
- Gepts P, Osborn TC, Rashka K, Bliss FA. 1986. Phaseolin-protein variability in wild forms and landraces of the common bean (Phaseolus vulgaris): evidence for multiple centers of domestication. Econ Bot 40: 451-468.Gepts P. 1990. Biochemical evidence bearing on the domestication of Phaseolus beans. Econ Bot 44(3S): 28-38.
- Gepts P, Debouck DG. 1991. Origin, domestication, and evolution of the common bean, Phaseolus vulgaris. In: Voysest O, Van Schoonhoven A (eds.), Common beans: research for crop improvement. CAB, Oxon, UK: pp. 7-53.
- González AM, Yuste-Lisbona FJ, Rodiño AP, De Ron AM, Capel C, García-Alcazar M, Lozano R, Santalla M. 2015. Uncovering the genetic architecture of Colletotrichum lindemuthianum resistance through QTL mapping and epistatic interaction analysis in common bean. Front. Plant Sci (DOI: 10.3389/fpls.2015.0014).
- Islam FMA, Basford KE, Jara C, Redden RL, Beebe S. 2002. Seed compositional and disease resistance differences among gene pools in cultivated common bean. Genet. Resour. Crop. Evol. 49: 285–293.
- Kelly JD, Gepts P, Miklas PN, Coyne DP. 2003. Tagging and mapping of genes and QTL and molecular marker-assisted selection for traits of economic importance in bean and cowpea. Field Crops Res 82: 135–154.
- Kelly JD. 2010. The Story of Bean Breeding. White paper prepared for BeanCAP & PBG Works on the topic of dry bean production and breeding research in the U.S. Michigan State University.
- Koenig R, Gepts P. 1989. Allozyme diversity in wild Phaseolus vulgaris: further evidence for two major centers of diversity. Theor Appl Genet 78: 809-817.
- Kwak M, Velasco D, Gepts P. 2008. Mapping homologous sequences for determinacy and photoperiod sensitivity in common bean (Phaseolus vulgaris). J Hered 99: 283-291.
- Lavin M, Herendeen PS, Wojciechowski MF. 2005. Evolutionary rates analysis of Leguminosae implicates a rapid diversification of lineages during the Tertiary. System. Biol. 54: 530–549.
- Matsuoka Y, Vigouroux Y, Goodman MM, Sanchez GJ, Buckler E, Doebley J. 2002. A single domestication for maize shown by multilocus microsatellite genotyping. Proc. Natl. Acad. Sci. USA 99: 6080–6084.
- McClean PE, Kami J, Gepts P. 2004. Genomics and genetic diversity in common bean. In: RF Wilson, HT Stalker, EC Brummer (Eds.) Legume Crop Genomics. AOCS Press, Champaign, Illinois, USA, pp. 60–82.
- McClean PE, Mamidi S, McConnell M, Chikara S, Lee R. 2010. Synteny mapping between common bean and soybean reveals extensive blocks of shared loci. BMC Genomics 11:184.
- Miklas PN, Kelly JD, Beebe SE, Blair MW. 2006. 2006. Common bean breeding for resistance against biotic and abiotic stresses: From classical to MAS breeding. Euphytica 147: 105–131.
- Porch TG, Beaver JS, Debouck DG, Jackson SA, Kelly JD, Dempewolf H. 2013 Use of Wild Relatives and Closely Related Species to Adapt Common Bean to Climate Change. Agronomy 3: 433-461.
- Powell W, Machray GC, Provan J. 1996. Polymorphism revealed by simple sequence repeats. Trends in Plant Science (1): 215-222.
- Repinski S, Kwak M, Gepts P. 2012. The common bean growth habit gene PvTFL1y is a functional homolog of Arabidopsis TFL1. Theor Appl Genet 124: 1539-154.
- Santalla M, Rodiño AP, De Ron AM. 2002. Allozyme evidence supporting southwestern Europe as a secondary center of genetic diversity for common bean. Theoretical Applied Genetics 104: 934-944.
- Santalla M, Lema M, Rodiño AP, González AM, Monteagudo AB, De Ron AM. 2005. Improvement of large-seeded common bean cultivars under sustainable cropping systems in Spain. Euphytica 142: 85-95.
- Schmutz J, McClean P, Mamidi S, Wu G, Cannon S, Grimwood J, Jenkins J, Shu S, Song Q, Chavarro C, Torres-Torres M, Geffroy V, Moghaddam SM, Gao D, Abernathy B, Barry K, Blair M, Brick MA, Chovatia M, Gepts P, Goodstein DM, Gonzales M, Hellsten U, Hyten DL, Jia G, Kelly JD, Kudrna D, Lee R, Richard MMS, Miklas PN, Osorno JM, Rodrigues J, Thareau V, Urrea CA, Wan M, Yu Y, Zhang M, Wing RA, Cregan PB, Rokhsar DS, Jackson SA. 2014. A reference genome for common bean and genome-wide analysis of dual domestications. Nature Genetics 46:707-713.
- Singh SP, Gepts P, Debouck DG. 1991. Races of common bean (Phaseolus vulgaris L., Fabaceae). Econ Bot 45: 379-396.Summerfield y Roberts, 1985
- Tohme J, González DO, Beebe S, Duque MC. 1996. AFLP analysis of gene pools of a wild bean core collection. Crop Sci. 36: 1375–1384.
- Quiroga G, González AM, Yuste F, Weller J, Sussmilch FC, Lozano R, Santalla M. 2015a. Identification and phylogenetic analysis of FT genes in common bean (Phaseolus vulgaris L.). Page 158. Eucarpia International Symposium on Protein Crops. Pontevedra, Spain.
- Quiroga X, González AM, Lores M, Capel J, Lozano R, Santalla. 2015b. Development of a new TILLING population of common bean. Page 159. Eucarpia International Symposium on Protein Crops. Pontevedra, Spain.
- Vlasova A, Capella-Gutiérrez S, Rendón-Anaya M, Hernández-Oñate M, Minoche A, Erb I, Câmara F, Prieto-Barja P, Corvelo A, Sanseverino W, Westergaard G, Dohm JC, Pappas GJ, Saburido-Alvarez S, Kedra D, Gonzalez I, Cozzuto L, Gómez-Garrido J, Aguilar-Morón MA, Andre N, Aguilar OM, Garcia-Mas J, Zehnsdorf M, Vázquez MP, Delgado-Salinas A, Delaye L, Lowy E, Mentaberry A, Vianello-Brondani RP, García JL, Alioto T, Sánchez F, Himmelbauer H, Santalla M, Notredame C, Gabaldón T, Herrera-Estrella A, Guigó R. 2016. The genome and transcriptome analysis of the Mesoamerican common bean and the role of gene duplications in establishing tissue and temporal specialization of genes. Genome Biology (DOI: http://dx.doi.org/10.1186/s13059-016-0883-6).
- Xu DH, Gai Y. 2003. Genetic diversity of wild and cultivated soybeans growing in China revealed by RAPD analysis. Plant Breeding 122: 503–506.
- Yuste-Lisbona FJ, Santalla M, Capel C, García-Alcázar M, De la Fuente M, Capel J, De Ron AM, Lozano R. 2012. Marker-based linkage map ofAndean common bean (Phaseolus vulgaris L.) and mapping of QTLs underlying popping ability traits. BMC Plant Biology 12 (136): 2-16.
















