Aplicación de la biotecnología para la detección de patógenos de patata
La variabilidad genética existente en los principales patógenos de la patata dificulta la detección visual de los síntomas de la enfermedad tanto en campo como en condiciones de cultivo controladas. Por ello, es necesario implementar técnicas de diagnóstico y detección que permitan su diferenciación e identificación de forma sencilla, rápida y precisa. El objetivo de este artículo es la descripción de algunos métodos de diagnóstico e identificación de virus, nematodos y mildiu de la patata, mediante la aplicación de técnicas moleculares.
El objetivo del diagnóstico es identificar lo antes posible patógenos para evitar el desarrollo de enfermedades que mermen la producción de un cultivo o lo hagan inservible. El diagnóstico molecular, al igual que todo proceso de diagnóstico, pretende la determinación de la causa de una enfermedad infecciosa. Sin embargo, a diferencia de un diagnóstico microbiológico convencional, el diagnóstico molecular va más allá ya que identifica parte del ADN o ARN del patógeno.
Los métodos moleculares están basados en la detección de parte del genoma del patógeno. Para poder desarrollar un método molecular debe conocerse parte del genoma o el genoma completo del microorganismo que se pretende detectar, ya sea un virus, un viroide, un hongo, un nematodo o una bacteria. Los métodos moleculares más importantes que se utilizan hoy en día en el diagnóstico de enfermedades infecciosas son la reacción en cadena de la polimerasa (PCR o PCR-TR) y la secuenciación. Estos métodos son directos y nos indican la presencia o ausencia del material genético del patógeno. A diferencia de otros métodos, no nos indican si ese patógeno está vivo o no. Por ello, en ocasiones no es suficiente confirmar la presencia del mismo, sino que debemos complementar estas pruebas con otras que nos indiquen la viabilidad del microorganismo.
1. Detección de virus de patata
Existen una gran cantidad de virus que infectan a la patata (Solanum tuberosum L.) y especies afines. Estos se clasifican en graves (virus Y y virus del enrollado o PLRV), que producen una deformación permanente de las hojas y pérdidas de cosecha del 30-50%, y virus leves, que normalmente producen síntomas más ligeros con pérdidas del 5 al 10%.

Los virus graves y leves (Foto 1) se perpetúan a través de tubérculos infectados y se transmite de forma eficiente a través de áfidos. El máximo porcentaje de infección para virus graves permitido en semilla de patata certificada varía entre el 8% (clase A) y el 10% (clase B), siendo sustancialmente más bajo en generaciones base y prebase. El PLRV es del tipo del género Polerovirus, con partículas isométricas de 25-30 nm y genoma monopartito de ARN unicatenario de sentido positivo de 5300-5700 nucleótidos. Son virus limitados al floema y transmitidos por pulgones de modo no persistente. Los virus leves (X, S, A, M y V) aún siendo taxonómicamente diversos, comparten ciertas características. El PVX y los carlavirus (PVS y PVM) comparten un elevado título en planta. Dos virus leves, en combinación, pueden dar lugar a síntomas graves como consecuencia de un sinergismo entre ellos, así la combinación PVA + PVX se conoce como crinckle y es similar al mosaico rugoso causado por PVY.
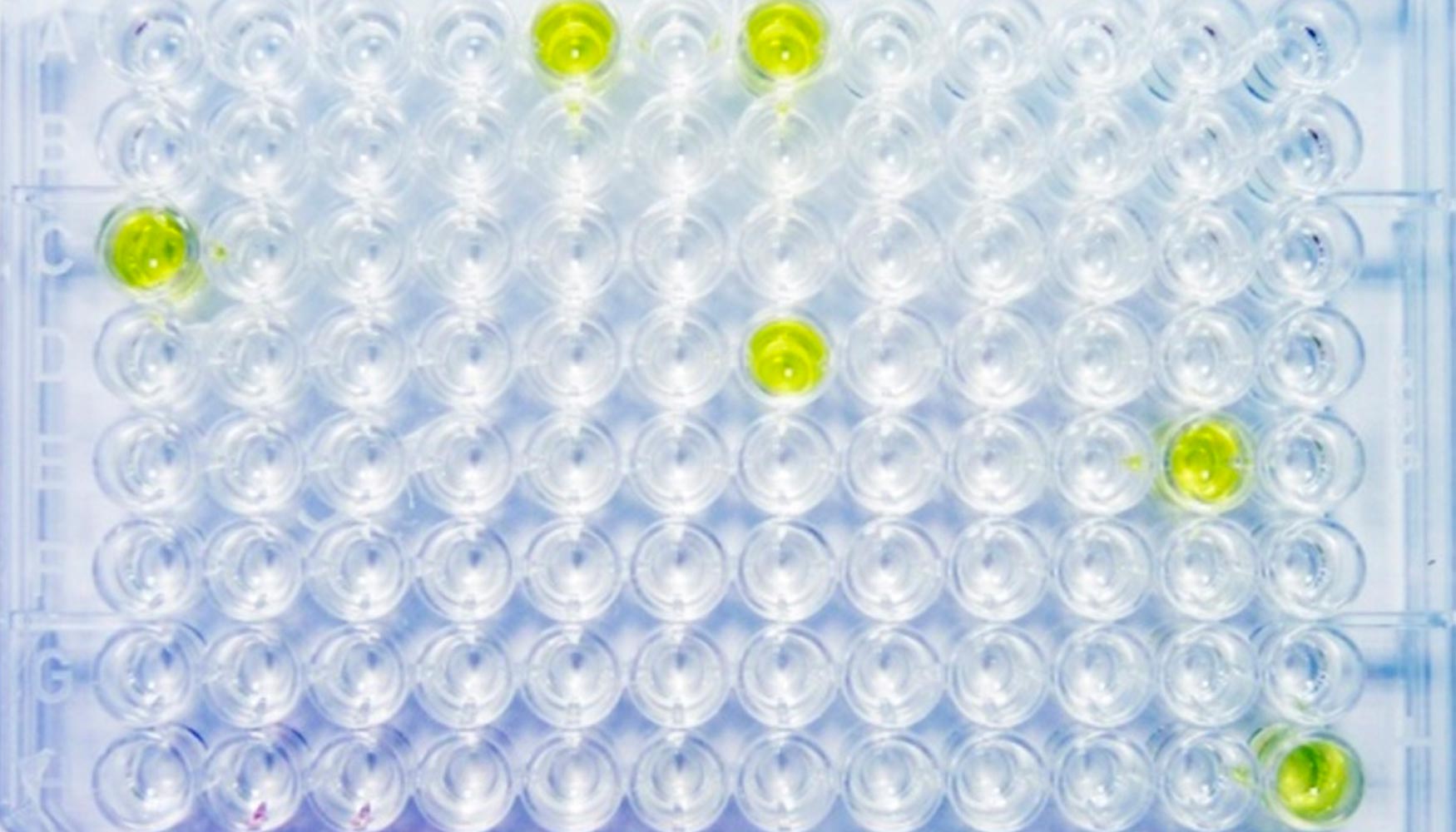
El ensayo inmunoabsorbente directo ligado a enzimas (ELISA) es una técnica inmuno-enzimática que permite la rápida detección de estos virus mediante reacciones antígeno-anticuerpo a través del producto generado (Foto 2). Actualmente existen numerosas variantes de la prueba ELISA que ofrecen distinto grado de sensibilidad y especificidad, pudiendo ser ELISA directo o indirecto en función de si se utiliza un único anticuerpo o dos (uno reacciona con el antígeno vírico y uno secundario marcado que reacciona con el anticuerpo primario); ELISA Sandwich, si utiliza dos anticuerpos que reaccionan con el antígeno (ELISA-DAS y ELISA-HADAS) o ELISA competitivo, si existe competición entre el anticuerpo de la muestra y el conjugado por los sitios de unión con el antígeno. A pesar de ser una prueba robusta y ampliamente utilizada para el diagnóstico de virus en patata, el ensayo ELISA presenta una serie de inconvenientes que limitan su eficacia. En este sentido, algunos autores han señalado su incapacidad de discriminar las cepas necróticas, la posibilidad de que algunas cepas recombinantes de PVY no sean detectadas o sean identificadas de forma incorrecta, la necesidad de contar con tubérculos brotados para detectar cantidades apreciables de proteínas virales (sensibilidad relativamente baja), así como la aparición de falsos positivos.
Para superar estas limitaciones se han puesto a punto diferentes técnicas moleculares aptas para diferenciar cepas virales basadas en la RT-PCR (Foto 3). Esta constituye una técnica prometedora por su versatilidad y potencial analítico cuyo uso no ha dejado de crecer durante los últimos años.
No obstante, su utilización exitosa en el campo del diagnóstico e identificación de virosis requiere de la obtención de ARN de gran calidad y la optimización de las condiciones de la PCR, requisitos que son costosos o difícilmente alcanzables para algunos laboratorios de análisis.

Para solucionar este problema, han sido desarrolladas nuevas variantes de la RT-PCR que incorporan un paso previo de captura específica o inespecífica con el objetivo de concentrar los ácidos nucleícos virales, incrementando así la especificidad y la sensibilidad de los ensayos, reduciendo sus costes y simplificando los procedimientos. Las principales variantes de la RT-PCR son la IC-RT-PCR, la DB-RT-PCR, la PC-RT-PCR/SC-RT-PCR y la SC-RT-PR, que resultan especialmente sensibles y eficientes en la discriminación de cepas de virus.
La denominada IC-RT-PCR o inmunocaptura seguida de reacción en cadena de la polimerasa tras transcripción inversa es una técnica híbrida que consiste en un primer paso de enriquecimiento de la muestra a través de la aplicación de inmunocaptura (fijación específica del antígeno de interés a un anticuerpo sobre una superficie determinada), seguida por una fase de transcripción inversa (generación de una cadena de ADN a partir de ARN) y PCR (reacción en cadena para la obtención de múltiples fragmentos de secuencias de ADN específicas a partir de las cuales es posible discriminar distintas cepas virales). Se trata de una técnica particularmente sensible y específica debido a que a la especificidad de la RT-PCR hay que sumar la propia de la inmunocaptura previa (aproximadamente diez veces más sensible que la RT-PCR). Otras como la DB-RT-PCR o unión directa seguida de reacción en cadena de la polimerasa tras transcripción inversa (DB-RT-PCR) es una variante de la RT-PCR cuya particularidad es que previamente a las fases de transcripción inversa y PCR, se produce una unión no específica de los virus a un soporte sólido en tubos de polipropileno. Esta técnica presenta una menor sensibilidad que la IC-RT-PCR. La PC-RT-PCR o captura de huella seguida de reacción en cadena de la polimerasa tras transcripción inversa (PC-RT-PCR) es un ensayo que se caracteriza por requerir una fase de captura de proteínas víricas en un soporte sólido utilizando agentes de captura no específicos de PVY anterior a realización de la RT-PCR (utilizando membranas de nylon o papel). Se trata de una técnica particularmente interesante por su simplicidad y su sensibilidad intermedia entre las técnicas DB-RT-PCR e IC-RT-PCR. Otra técnica como la SC-RT-PCR o captura con gel de sílice seguida de reacción en cadena de la polimerasa tras transcripción inversa es otro método basado en la técnica de RT-PCR que incluye un primer paso de captura de ácidos nucleicos virales en gel de sílice. Esta técnica, constituye una alternativa altamente sensible y eficiente.
2. Identificación de especies de nematodo del quiste en patata
Las dos especies de nematodos del quiste de la patata (PCN), Globodera pallida (Stone) Behrens y Globodera rostochiensis (Wollenweber) Behrens, están reconocidas como organismos nocivos para las patatas. Las disposiciones de la Directiva 2007/33/CE del Consejo, de 11 de junio de 2007, relativa al control de los nematodos del quiste de la patata han tenido en cuenta la necesidad de exámenes oficiales que garanticen la ausencia de nematodos del quiste de la patata en parcelas en las que se planten o se almacenen patatas de siembra destinadas a la producción de patatas de siembra y determinados vegetales destinados a la producción de vegetales para la plantación. En aquellas parcelas donde se detecte la presencia de nematodo del quiste, una de las formas de lucha reconocida por la normativa es el uso de variedades de patata resistentes. Estas variedades resistentes actúan de manera que al ser sembradas y comenzar a producir raíces, emiten señales químicas que los nematodos son capaces de percibir y salen de los quistes para comenzar un nuevo ciclo de vida. Cuando las larvas de las hembras intentan adherirse a las raíces de las variedades resistentes, estas no permiten que completen su ciclo, y se produce un descenso importante en la población del parásito.
De las variedades disponibles en el mercado internacional, ninguna tiene resistencia completa a las dos especies y a todos sus patotipos. Citamos algunas interesantes comercialmente para nuestros agricultores: Agria, resistente a G. rostochiensis, patotipo Ro1; Innovator: Resistente a G. pallida, patotipos Pa2 y 3; Santé: Resistente a G. rostochiensis, patotipos Ro1, 2, 3 y 4 y a G. pallida, patotipo Pa2, con resistencia moderada a Pa1 y 3. El uso de variedades resistentes es muy eficaz, siempre que conozcamos cual es la especie y el patotipo de nematodo presente en la parcela. Si utilizamos una variedad resistente a la especie o patotipo en una parcela donde la especie o patotipo es distinto, lo que haremos es multiplicar la población del parásito en vez de disminuirla. Por ello, es imprescindible conocer la población presente en cada parcela. Ahí es donde entran los métodos moleculares, que nos permiten diferenciar con facilidad la especie presente. Para ello, una vez obtenidos los quistes de la parcela, se extrae el ADN de los huevos que están dentro los quistes. El ADN se añadirá a una mezcla de reactivos en la que se incluirán tres primers o secuencias iniciadoras que nos permitirán multiplicar exponencialmente una parte del genoma de cada una de las especies, si están presentes.
- ITS5: 5´-GGA AGT AAA AGT CGT AAC AAG G-3´
- PITSr3: 5´-AGC GCA GAC ATG CCG CAA-3´ Específico de G. rostochiensis
- PITSp4: 5´-ACA ACA GCA ATC GTC GAG –3´ Específico de G. pallida
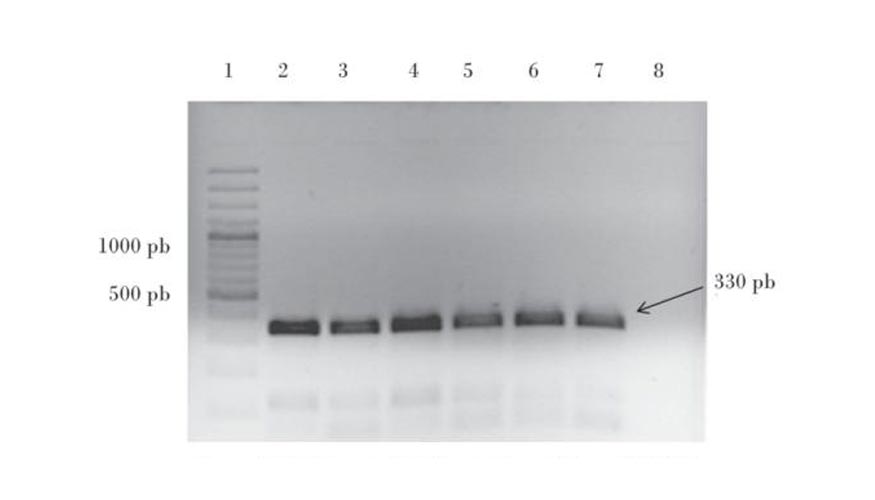
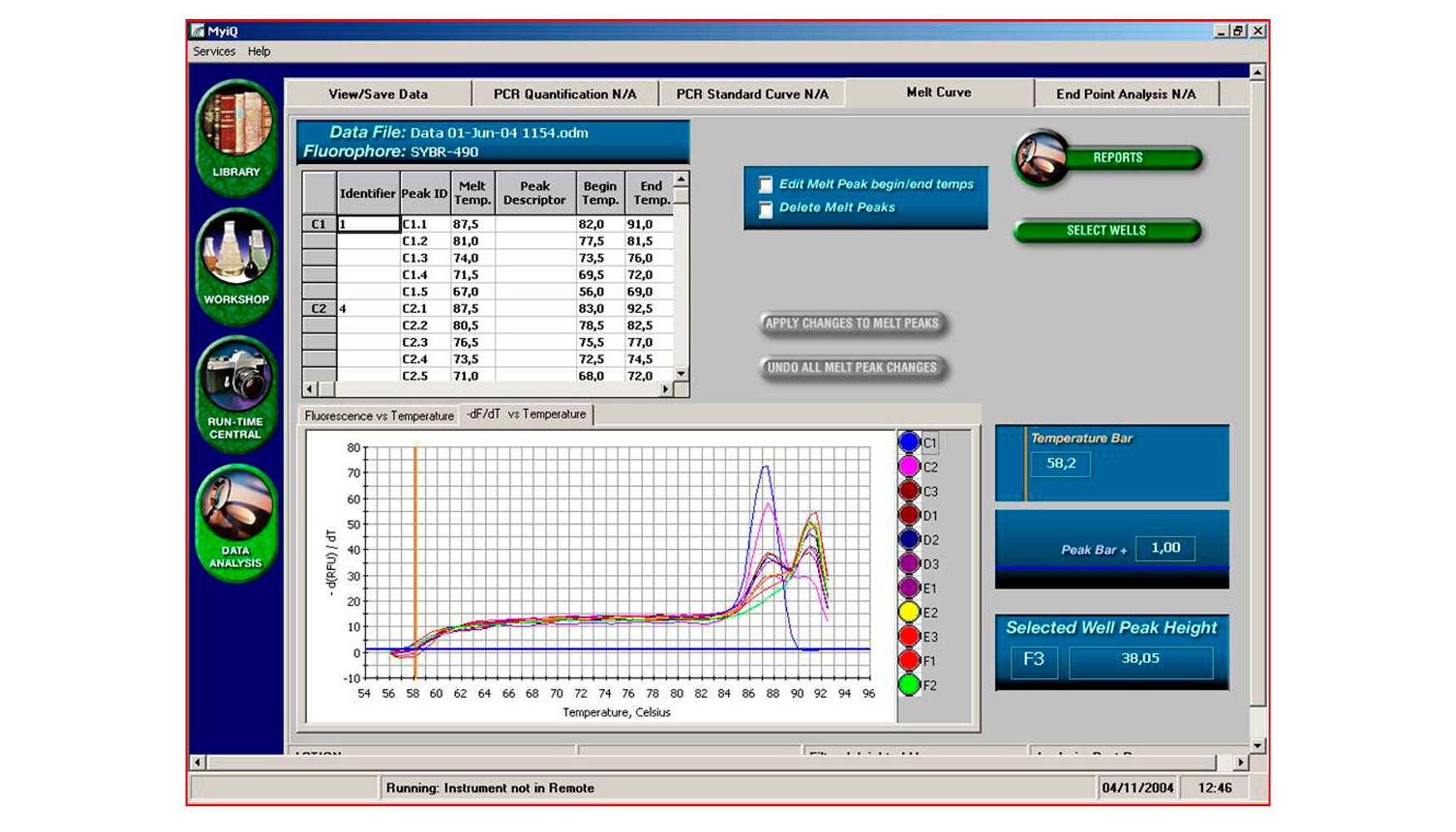
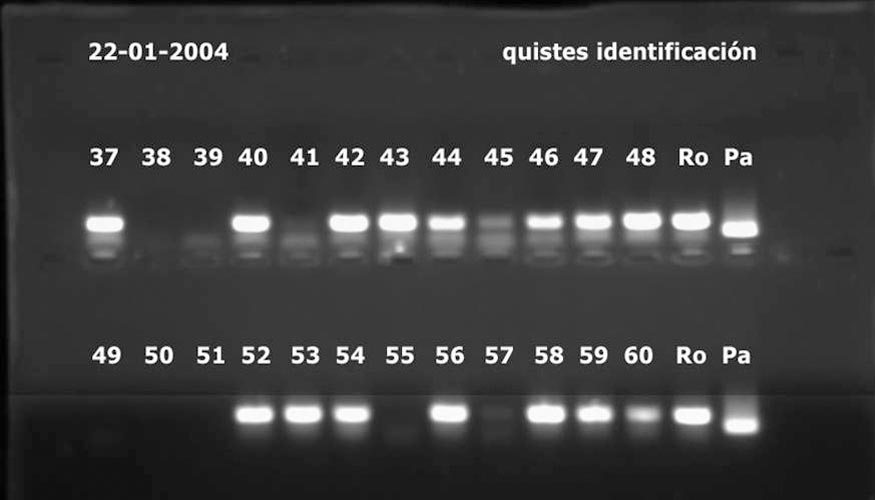
La mezcla se colocará en un equipo de PCR que mediante cambios precisos de temperatura, permitirá la multiplicación de segmentos específicos de una u otra especie. Según el tipo de técnica que utilicemos, el resultado será en forma de gráfico que captará fluorescencia (PCR-RT) (Fotos 5) o en forma de bandas si se trata de PCR convencional (Fotos 6).
3. Caracterización del mildiu de la patata
El Oomycete Phytophthora infestans (Mont.) de Bary, es una especie de distribución mundial, causante del mildiu o tizón tardío, una de las enfermedades más limitantes y devastadoras de la patata aunque también puede afectar a otras Solanáceas, como el tomate (S. lycopersicum) y el pepino dulce (S. muricatum Ait.). El pseudohongo crece en el tejido de la planta formando hifas a modo de ramas en las que se producen los esporangios. Estos tienen forma de pequeños limones que se desprenden y a través del viento y el agua son transportados para colonizar otras plantas. Cuando los esporangios encuentran tejido sano, germinan y comienzan un nuevo ataque. La caracterización molecular de las razas existentes es de gran importancia para el estudio de la evolución del patógeno y poder optimizar los métodos de control de la enfermedad. El patógeno presenta dos tipos de reproducción: sexual y asexual. Para que se produzca la reproducción sexual se debe comportar como una especie heterotálica, es decir, necesita la existencia dos tipos de razas con apareamientos diferentes (A1 y A2). La unión de los anteridios (gametos masculinos) y oogonios (gametos femeninos), produce una oospora diploide, la cual por recombinación genética puede generar una mayor variabilidad en patogenicidad y agresividad. La formación de estas oosporas le permite a P. infestans sobrevivir en condiciones adversas del invierno, períodos secos y ausencia de hospedantes durante meses o incluso años.
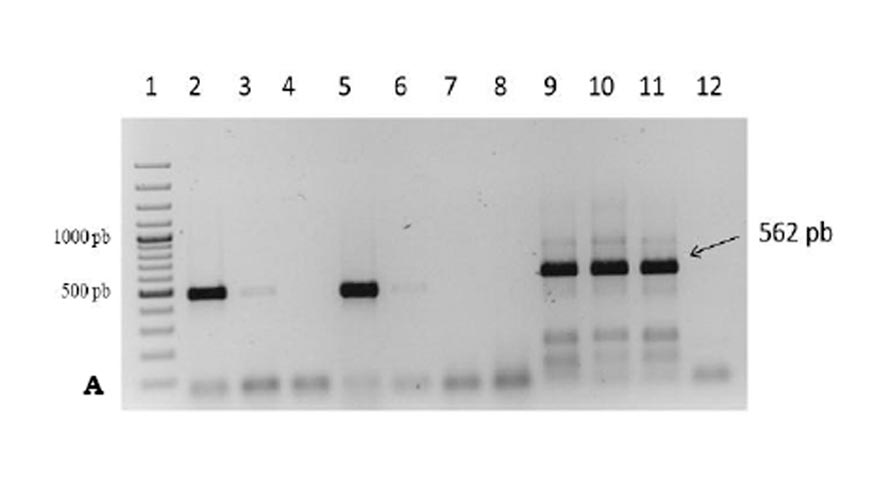
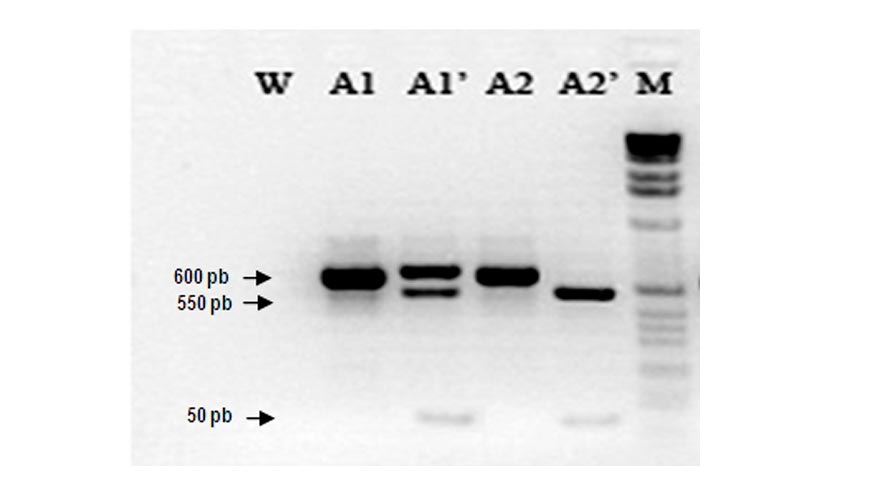
La determinación del apareamiento por métodos moleculares se realiza como medida de confirmación de los diferentes aislados existentes se realiza mediante caracterización del ADN. Para ello se lava el micelio, se liofiliza y se tritura con nitrógeno líquido. La extracción del ADNse realiza mediante un kit comercial, para luego realizar la amplificación mediante la técnica PCR con el fin de determinar la presencia o ausencia de segmentos específicos (locus) del genoma que caracterizan individuos del tipo de apareamiento A1 y A2. Esto se visualiza mediante la aparición en un gel de agarosa de fragmentos específicos (bandas) que caracterizan a cada tipo de apareamiento. Los marcadores moleculares específicos son de tipo CAPs (Cleaved Amplified Polymorphic Sequence) con los cebadores W16-1 y W16-2, previa digestión con la endonucleasa de restricción HaeIII que permite observar en un mismo gel de agarosa ambos tipos sexuales (A1 y A2). El tipo de apareamiento A1 muestra tres bandas patrón con un tamaño molecular de 600, 550 y 50 pb, mientras que para el tipo de apareamiento A2 presenta dos fragmentos de restricción con bandas de tamaño molecular de 550 y 50 pb (Foto 7).
Además de identificar el tipo de apareamiento de los aislados la aplicación de la técnica PCR con marcadores específicos sirve para poder caracterizar los diferentes genotipos del patógeno (Figura 1). El método consiste en la extracción del ADN a partir de placas Petri conteniendo el medio de Agar-centeno en el que fueron cultivados los aislados del pseudohongo. Para ello se utiliza un kit comercial. Seguidamente se emplean 12 marcadores microsatélites SSR polimórficos descritos por varios autores. La amplificación se realiza por PCR multiplex en un termociclador.
Agradecimientos
Este trabajo ha sido financiado en el marco del proyecto RTA2013-00006-C03-01 y por el Gobierno Vasco.
Referencias
- Cooke, D. E. L., and Andersson B. 2013. Phytophthora infestans and potato Late Blight in Europe En: Phytophthora: A Global Perspective. Ed. K. Lamour. pp. 59-67.
- Directiva 2007/33/CE del Consejo, de 11 de junio de 2007, relativa al control de los nematodos del quiste de la patata. L256/12-22.
- Gil J.F., Cotes J.M., Marin M. 2011. Incidencia y caracterización molecular del Potato Leafroll Virus (PLRV) en las principales regiones productoras de papa de Colombia. Fitosanidad 15:1-3.

